|
 |
| Res. Plant Dis > Volume 27(2); 2021 > Article |
|
요 약
2019년 6월 충남 논산의 당귀 재배 농가에서 원형반점, 괴사반점, 황화, 모자이크 등의 증상을 보이는 당귀잎을 채집하였고, 이들 시료에 대한 바이러스 감염 여부를 확인하기 위하여 전자현미경 검경과 감염우려가 있는 바이러스 3종(cucumber mosaic virus, broad bean wilt virus 2, tomato spotted wilt virus [TSWV])에 대해 reverse transcription polymerase chain reaction 진단을 수행한 결과 TSWV에 대해 양성반응을 보였다. 당귀 TSWV의 생물학적 특성을 구명하기 위해 순수분리하여 5과 28종의 지표식물과 일당귀에 즙액접종하여 기주범위와 병원성을 확인하였다. TSWV의 3 segment (L, M, S)의 전체 염기서열을 결정하고 기존에 보고된 TSWV 분리주와 계통분석을 한 결과, 당귀 TSWV 분리주(NS-AG-28)는 논산지역의 머위 분리주(TSWV-NS-BB20)와 가장 유사한 것으로 나타났다. TSWV는 넓은 기주범위를 가지고 있고 특히 고추, 토마토 등 가지과 작물에 큰 피해를 주고 있기 때문에, 당귀가 TSWV의 중간기주로서 작용하지 않도록 주의하고, 당귀의 바이러스병 피해를 예방하기 위하여 지속적인 모니터링이 요구된다.
ABSTRACT
In June 2019, Angelica acutiloba plants showing virus-like symptoms such as chlorotic local lesion and mosaic on the leaves were found in a greenhouse in Nonsan, South Korea. To identify the causal virus, we collected 6 symptomatic A. acutiloba leaf samples and performed reverse transcription polymerase chain reaction (RT-PCR) analysis using specific detection primers for three reported viruses including tomato spotted wilt virus (TSWV). RT-PCR results showed that five symptomatic samples were positive for TSWV. Mechanical sap inoculation of one of the collected TSWV isolate (TSWV-NS-AG28) induced yellowing, chlorosis and mosaic symptoms in A. acutiloba and necrotic local lesions and mosaic in Solanaceae species. Phylogenetic analysis based on the complete genome sequences showed that TSWV-NS-AG28 had a maximum nucleotide identity with TSWV-NS-BB20 isolated from butterbur in Nonsan, South Korea. To our knowledge, this is the first report of TSWV infection in A. acutiloba.
당귀는 미나리과의 다년생 초본의 약용작물로 원산지에 따라 한국은 참당귀(Angelica gigas), 일본은 일당귀(A. acutiloba), 중국은 중국당귀(A. sinensis)로 나뉘어진다. 국내에서는 참당귀와 일당귀가 주로 재배되고 있으며, 참당귀는 주로 한약재로, 일당귀는 쌈채소로 이용된다. 당귀의 뿌리는 혈류를 촉진하고 빈혈을 치료하는 데 사용되며, 항암효과 및 혈압강하에 효능이 있다고 알려져 있다(Cho 등, 2017; Kim 등, 2011). 국내 재배면적은 2018년 기준으로 참당귀는 457 ha, 일당귀는 41 ha이고, 참당귀는 주로 강원도(260 ha)와 경북(146 ha)에서, 일당귀는 충남 (22 ha)과 경북(10 ha)에서 재배되고 있다(KOSIS, 2019).
당귀에 감염하는 바이러스로는 2009년 중국당귀에서 Japanese hornwort mosaic virus (JHMV; genus Potyvirus, family Potyviridae)가 처음 발견되었고, 최근에 이 바이러스에 대한 간편진단법이 보고되었다(Zhang 등, 2009, 2020). 국내에서는 모자이크 증상을 보인 당귀에서 cucumber mosaic virus (CMV; genus Cucumovirus, family Bromoviridae)가 검출되었으나(Rural Development Administration, 2018), 바이러스에 의한 피해는 거의 알려져 있지 않다.
토마토반점위조바이러스(tomato spotted wilt virus, TSWV)는 Orthotospovirus속의 대표적인 바이러스로서, 꽃노랑총채벌레(Frankliniella occidentalis)를 포함한 10여종의 총채벌레에 의해 전염하는 것으로 알려져 있다(Morse와 Hoddle, 2006). TSWV 유전자는 L segment (8.9 kb), M segment (4.8 kb)와 S segment (2.9 kb)의 세 가닥의 절편(segment)으로 구성되어 있다(Adkins, 2000). 기주 범위가 매우 넓어 약 1,000여종의 식물에 감염하는 것으로 알려져 있으며, 주요 피해 작물은 고추, 토마토 등 가지과 작물, 땅콩 등 콩과 작물 및 국화 등 화훼류에 큰 피해를 주고 있다(Pappu 등, 2009; Parrella 등, 2003). TSWV 피해증상은 작물의 잎과 과일에 원형반점, 괴사반점, 황화 및 모자이크 증상이 나타나며 심한 경우 식물체 전체가 고사된다.
국내에서는 2003년 충남 예산지역 파프리카 재배농가에서 처음으로 확인된 후(Kim 등, 2004), 현재 고추, 토마토, 상추, 국화 등의 작물에서 전국적으로 발생하고 있고, 특히 최근에는 구기자, 땅콩, 페페로미아 등 새로운 작물로도 확산되어 피해를 주고 있어 경제적 손실이 크다(Cho 등, 2020; Kwak 등, 2020; Yoon 등, 2019). 특히, 충남 논산지역에서는 최근 고추, 토마토 등에서 TSWV가 발생하면서 주변의 채소작물인 상추, 로메인상추 및 약용작물인 머위 등으로 확산되어 피해가 발생하였다(Kwak 등, 2018).
본 연구에서는 2019년 논산의 당귀 재배농가에서 잎에 황화반점, 엽맥퇴록 및 모자이크 등의 바이러스 증상을 보이는 당귀를 채집하였고, 당귀 잎으로부터 TSWV를 분리하여 전체 유전자 염기서열 분석 및 생물검정을 통하여 당귀에서 TSWV를 분류동정하였다.
바이러스 수집 및 증상. 2019년 6월 약용작물을 재배하는 농가의 당귀포장에서 황화반점, 엽맥퇴록 및 모자이크 등의 바이러스 병징을 보이는 개체가 발견되었다(Fig. 1). 당귀는 쌈채소로 이용되는 일당귀로 뿌리 증식을 통해 재배되고 있었다. 병징은 병이 진행됨에 따라 심하면 잎 전체가 퇴록되고 생육이 저하되었다. 바이러스의 정확한 동정을 위하여 병징이 있는 당귀 시료 6점을 채집하였다.
전체 RNA 추출 및 RT-PCR 진단. 유전자 진단에 앞서 채집한 당귀 잎 시료는 dip method에 의한 direct negative staining 법(Hitchborn and Hills, 1965)으로 염색한 후 투과전자현미경(transmission electron microscopy, LEO 912AB, Carl Zeiss, Jena, Germany)으로 바이러스 입자를 관찰한 결과, 막대형이나 사상형 등의 입자가 관찰되지 않아서 중국에서 알려진 사상형 바이러스인 JHMV는 감염되지 않은 것으로 확인되었다. 감염 바이러스의 정확한 진단을 위해 total RNA를 추출하고(Plant RNA Prep Kit, Biocubesystem, Suwon, Korea), 기존에 알려진 CMV 및 기주범위가 넓고 약용작물에 잘 감염하는 잠두위조 바이러스2(broad bean wilt virus 2, BBWV2) (Kwak 등, 2017; Kwon과 Seo, 2019; Kwon 등, 2016)와 논산지역에서 유행하고 있는 TSWV 등 3종의 바이러스 특이 프라이머(Table 1)를 이용하여 RT-PCR 진단하였다. 진단 결과, 1점을 제외한 모든 시료에서 TSWV가 검출되었다(Fig. 2).
기주범위 및 병원성 검정. 병원성 검정과 기주범위 조사를 위하여 TSWV에 감염된 당귀 잎 조직을 0.01 M phospate buffer (pH 7.0)에 마쇄하여 얻은 즙액을 Chenopodium quinoa에 접종하여 3차례에 걸쳐 단일병반(single local lesion)을 순수분리하고, Nicotiana tabacum cv. Samsun에서 증식하여 접종원으로 사용하였다. 순수분리된 TSWV 분리주 NS-AG28은 농업 생명자원으로 기탁하였다(기탁번호 CV191116-5). 당귀에서의 병원성을 확인하기 위하여 일당귀 유묘에 NS-AG28를 즙액접종한 결과 접종엽에서는 병징이 나타나지 않았고 상엽에서는 황화, 퇴록 및 모자이크 증상이 나타났다(Table 2, Fig. 3). 포장에서의 심한 황화반점의 증상과는 차이가 있었다. 기주범위를 조사하기 위하여 지표식물 5과 28종을 사용하여 3반복 수행한 후 접종엽과 상엽에서 병징을 관찰하였다(Table 2, Fig. 3). 생물 검정 결과 N. benthamiana를 포함한 18종에서 전신감염을 보였으며, 담배에서는 주로 접종엽에는 괴사증상과 원형반점을 보였고, 상엽에서 괴사증상과 모자이크 증상을 보였다. TSWV에 취약한 작물인 고추와 토마토의 경우, 고추에서는 접종엽에 원형반점과 괴사증상을 나타내었고 상엽에서는 모자이크 증상을 보였으며, 토마토에서는 접종엽에 증상이 없이 상엽에서 모자이크 증상을 나타내었다. N. tabacum cv. Xanthi 등 3종에서는 접종엽에서만 원형반점 및 괴사증상을 보이고 상엽에서는 병징이 나타나지 않았으며, 호박을 포함한 박과작물 4종과 강낭콩를 포함한 콩과작물 3종에서는 병징이 나타나지 않았다. 이러한 생물검정 결과는 RT-PCR 진단으로 최종 확인되었다.
염기서열 및 계통분석. 전체 유전자 염기서열 분석을 위하여 이전에 보고된 TSWV 특이 프라이머를 사용하여 RT-PCR과 클로닝을 수행하였고(Kwak 등, 2020), Bionics사(Bionics, Daejeon, Korea)에 의뢰하여 전체염기서열을 얻었다. DNA Star v. 5.02 (Lasergene, Madison, WI, USA)의 SeqMan 프로그램을 사용하여 DNA assembly를 수행하여 최종적으로 L (8,914 bp), M(4,751 bp), S (2,916 bp)의 염기서열을 결정하였고 NCBI의 GenBank에 등록하였다(Table 3). 기존에 NCBI GenBank에 보고되어 있는 TSWV 34개 분리주를 이용하여 각 segment의 전체 염기서열을 Geneious Pro 10 software를 이용하여 alignment를 수행하고 염기서열 상동성을 분석한 결과, 당귀 TSWV 분리주(TSWV-NS-AG28)는 국내 논산지역의 머위에서 분리된 TSWVNS-BB20 분리주와 99-100%의 가장 높은 상동성을 보였고, 스페인의 토마토에서 분리된 TSWV-LL-N.05 분리주와도 98% 이상의 높은 상동성을 보였다. 또한 MEGA 7 프로그램의 maximum likelihood method에 의한 계통분석 결과, TSWV는 크게 4개의 그룹으로 나뉘는데 각각의 segment에서 머위 TSWV 분리주와 같은 그룹에 속하는 것을 확인할 수 있었다(Fig. 4). 본 보고는 TSWV의 새로운 기주인 당귀에서의 TSWV 발생의 첫 보고이다. 당귀에서의 TSWV 피해를 막기 위해서, 또한 당귀는 고추, 토마토 등 주요 작물로의 TSWV 확산의 중간기주로 작용할 수 있기 때문에, 당귀 재배지역에서의 TSWV의 지속적인 모니터링이 필요할 것으로 보인다.
Acknowledgments
This research was supported by a grant from the Agenda Program (PJ01424001) funded by the Rural Development Administration of Korea.
Fig. 1.
Virus-like symptoms on the leaves of Angelica acutiloba. (A) Chlorotic and necrotic local lesion, and mosaic. (B) Chlorotic local lesion and vein chlorosis.

Fig. 2.
Agarose gel electrophoresis of reverse transcription polymerase chain reaction from symptomatic leaves (lanes 1, 2, 3, 4, 5, and 6) using the specific primers of CMV, BBWV2, and TSWV. CMV, cucumber mosaic virus; BBWV2, broad bean wilt virus 2; TSWV, tomato spotted wilt virus; N, negative; P, positive control; M, 100 bp ladder.
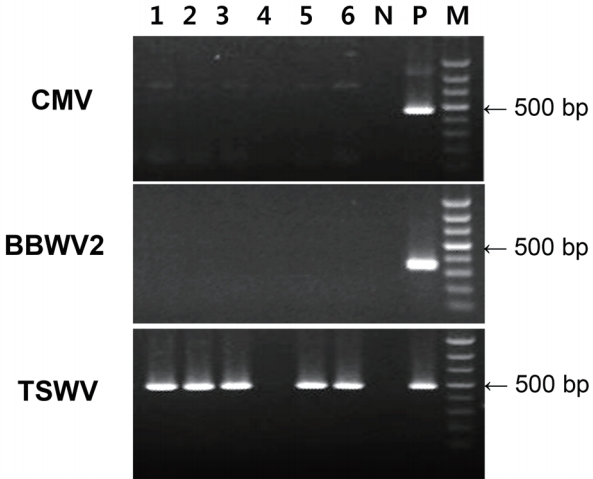
Fig. 3.
Symptoms on the indicator plants mechanically inoculated with TSWV isolate NS-AG28: (A) Angelica acutiloba, (B) Nicotiana benthamiana, (C) N. clevelandii, (D) N. glutinosa, (E) N. occidentalis, (F) N. rustica, (G) N. tabacum cv. ’Xanthi-nc’, (H) N. tabacum cv. Xanthi, (I) N. tabacum cv. Samsun, (J) N. tabacum cv. KY57, (K) N. tabacum cv. BY, (L) Solanum lycopersicum cv. Seogwang, (M) S. lycopersicum cv. Tenten, (N) Capsicum annuum cv. Quarri, (O) C. annuum cv. Supermainta, and (P) Petunia hybrida. TSWV, tomato spotted wilt virus.

Fig. 4.
Phylogenetic analysis based on the complete nucleotide sequences of (A) L segments, (B) M segments, and (C) S segments of TSWV to examine the evolutionary position of TSWS-NS-AG28 isolated from Angelica acutiloba. The phylogenetic tree was constructed by the maximum likelihood method using best fit nucleotide substitution models for L segments (TN93 + G), M segments (T92 + G), and S segments (T92 + G) with 1,000 bootstrap replications. TSWV, tomato spotted wilt virus.
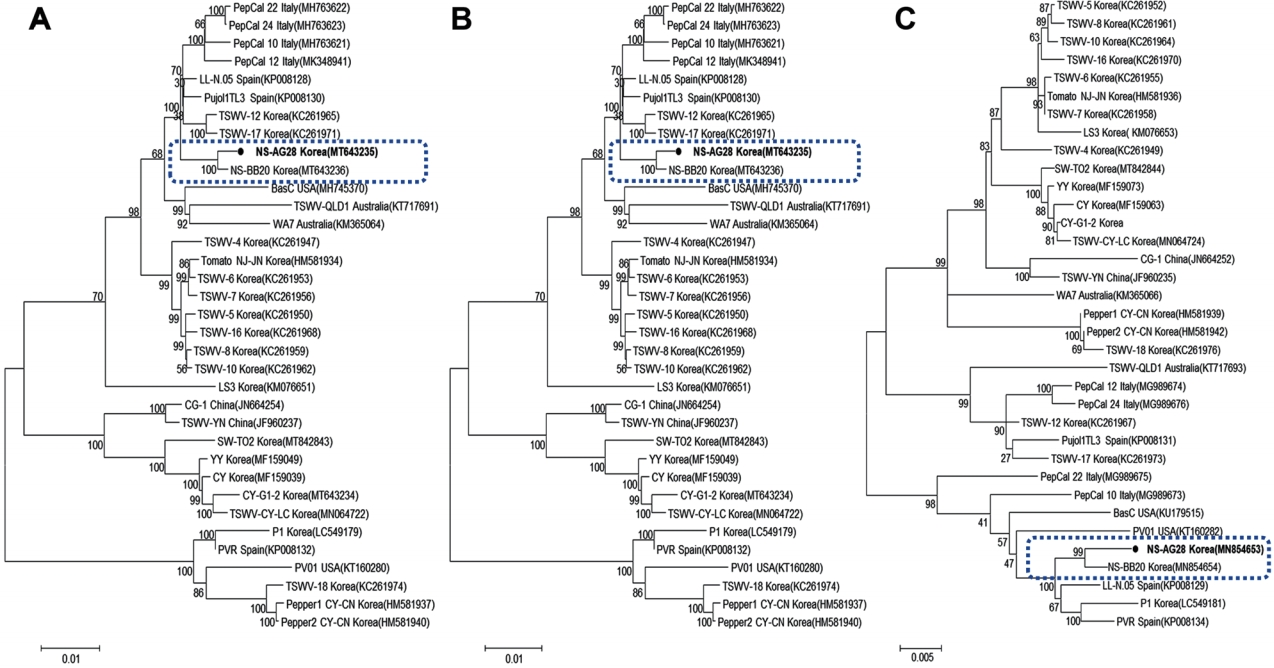
Table 1.
List of primers used to detect viruses in Angelica spp.
Table 2.
Symptoms observed on indicator plants mechanically inoculated with TSWV-NS-AG28 isolate from Angelica acutiloba
| Indicator plant | Symptomsa | TSWV incidenceb | |
|---|---|---|---|
| Apiaceae | Angelica acutiloba | -/cl, m, y | 1/1 |
| Solanaceae | Nicotiana benthamiana | -/m | 3/3 |
| N. clevelandii | nl/nl, m | 3/3 | |
| N. debneyi | nl/nl, m | 3/3 | |
| N. glutinosa | nl/nl, m | 3/3 | |
| N. occidentalis | nl/nl, m | 3/3 | |
| N. rustica | nl,m,rs/nl,m,rs | 3/3 | |
| N. tabacum cv. Xanthi-nc | nl,rs/nl,m,vn | 3/3 | |
| N. tabacum cv. Xanthi | rs/- | 3/3 | |
| N. tabacum cv. Samsun | rs, nl/m | 3/3 | |
| N. tabacum cv. KY57 | nl/nl,y,vn | 3/3 | |
| N. tabacum cv. BY | nl,rs/vn | 3/3 | |
| Solanum lycopersicum cv. Seogwang | -/m | 3/3 | |
| S. lycopersicum cv. Tenten | -/m | 2/3 | |
| Capsicum annuum cv. Quarri | rs,nl/m | 3/3 | |
| C. annuum cv. Supermainta | rs/m | 2/3 | |
| Petunia hybrida | nl/nl | 3/3 | |
| Datura stramonium | nl/nl,m,rs,dist | 3/3 | |
| Physalis angulata L. | nl/- | 0/3 | |
| Leguminosae | Phaseolus vulgaris | -/- | 0/3 |
| Pisum sativum | -/- | 0/3 | |
| Vigna unguiculata | -/- | 0/3 | |
| Chenopodiaceae | Chenopodiumamaranticolor | nl/- | 3/3 |
| C. quinoa | nl/nl | 3/3 | |
| Cucurbitaceae | Cucurbita moschata | -/- | 0/3 |
| Cucumis melo | -/- | 0/3 | |
| C. sativus | -/- | 0/3 | |
| Citrullus lanatus | -/- | 0/3 | |
| Amaranthaceae | Gomphrena globosa | nl/dist | 3/3 |
Table 3.
Hosts, origins, and accession numbers of TSWV isolates
References
Adkins, S. 2000. Tomato spotted wilt virus: positive steps towards negative success. Mol. Plant Pathol. 1: 151-157.


Cho, N. J., Lee, W. H., Kim, K. K. and Han, H. S. 2017. Investigation of the antioxidant effect of Angelicae Radix from Korea, China and Japan. J. Physiol. Pathol. Korean Med. 31: 182-187.

Cho, S.-Y., Kim, S.-M., Kim, S. and Lee, B. C. 2020. First report of tomato spotted wilt virus infecting Arachis hypogaea in Korea. J. Plant Pathol. 102: 271.

Hitchborn, J. H. and Hills, G. J. 1965. The use of negative staining in the electron microscopic examination of plant viruses in crude extracts. Virology 27: 528-540.


Kim, J.-H., Choi, G.-S., Kim, J.-S. and Choi, J.-K. 2004. Characterization of tomato spotted wilt virus from paprika in Korea. Plant Pathol. J. 20: 297-301.

Kim, M., Kang, H.-J., Kwak, H.-R., Kim, J.-E., Kim, J., Seo, J.-K. et al. 2017. First report of impatiens necrotic spot virus in Hoya carnosa in Korea. Res. Plant Dis. 23: 383-387. (In Korean)

Kim, S. A., Oh, H. K., Kim, J. Y., Hong, J. W. and Cho, S. I. 2011. A review of pharmacological effects of Angelica gigas, Angelica sinensis, Angelica acutiloba and their bioactive compounds. J. Korean Orient. Med. 32: 1-24.
KOSIS. 2019. Korea Statistical Information Servie. URL https://kosis.kr [30 May 2021]
Kwak, H.-R., Go, W.-R., Kim, J.-E., Park, G., Baek, E., Kim, M. et al. 2018. Spread of tomato spotted wilt orthotospovirus into various vegetables in Nonsan area of Korea. In: Proceedings of 2018 Annual Meeting on the Korean Society of Pesticide Science, ed. by The Korean Society of Pesticide Science. pp. 319The Korean Society of Pesticide Science, Suwon, Kore. pp. a pp.
Kwak, H.-R., Go, W.-R., Kim, M., Kim, C.-S., Choi, H.-S., Seo, J.-K. et al. 2017. First report of broad bean wilt virus 2 in Gynura procumbens in Korea. Plant Dis. 101: 514.

Kwak, H.-R., Kim, M.-K., Nam, M., Kim, J.-S., Kim, K.-H., Cha, B. et al. 2013. Genetic compositions of broad bean wilt virus 2 infecting red pepper in Korea. Plant Pathol. J. 29: 274-284.



Kwak, H.-R., Son, S.-W., Choi, H.-Y., Go, W.-R., Kim, J.-E., Baek, E. et al. 2020. First report of tomato spotted wilt virus in Lycium chinense. Australas. Plant Dis. Notes. 15: 5.

Kwon, S.-J., Cho, I.-S., Yoon, J.-Y., Choi, S.-K. and Choi, G.-S. 2016. First report of broad bean wilt virus 2 in Dioscorea opposita Thunb. in Korea. Plant Dis. 100: 538.

Kwon, S.-J. and Seo, J.-K. 2019. First report of broad bean wilt virus 2 in Achyranthes bidentata in Korea. Plant Dis. 103: 165.

Pappu, H. R., Jones, R. A. C. and Jain, R. K. 2009. Global status of tospovirus epidemics in diverse cropping systems: successes achieved and challenges ahead. Virus Res. 141: 219-236.


Parrella, G., Gognalons, P., Gebre-Selassiè, K., Vovlas, C. and Marchoux, G. 2003. An update of the host range of tomato spotted wilt virus. J. Plant Pathol. 85: 227-264.
Rural Development Administration. 2018. Diagnosis Method for Newly Developed Angelica acutiloba Mosaic Symptom. Rural Development Administration, Suwon, Korea.
Yoon, J. Y., Choi, G. S., Kwon, S. J. and Cho, I. S. 2019. First report of tomato spotted wilt virus infecting Peperomia obtusifolia in South Korea. Plant Dis. 103: 593.

- TOOLS
-
METRICS

- Related articles
-
First Report of Leaf Spot in Fischer’s Ragwort Caused by Didymella ligulariae2023 March;29(1)
First Report of Tomato Spotted Wilt Virus in Oxypetalum coeruleum in Korea2022 December;28(4)
First Report of Tomato Spotted Wilt Virus on Iris domestica in South Korea2021 March;27(1)
First Report of Black Rot Caused by
Diplodia seriata on Apple2018 December;24(4)



 PDF Links
PDF Links PubReader
PubReader ePub Link
ePub Link Full text via DOI
Full text via DOI Download Citation
Download Citation Print
Print