Pseudomonas syringae pv. syringae에 의한 애호박 세균점무늬병
Bacterial Spot Disease of Green Pumpkin by Pseudomonas syringae pv. syringae
Article information
Abstract
A pathogen that causes a new disease on green pumpkin in the nursery and the field was characterized and identified. Symptoms of the disease on green pumpkin were water soaking lesions and spots with strong yellow halo on leaf, brown lesion on flower, and yellow spot on fruit. The bacterial isolates from the leaf spot were pathogenic on the 8 curcubitaceae crop plants, green pumpkin, figleaf gourd, wax gourd, young pumpkin, zucchini, cucumber, melon, and oriental melon, whereas they did not cause the disease on sweet pumpkin and watermelon. They were Gram-negative, rod shape with polar flagella, fluorescent on King’s B agar and LOPAT group 1a by LOPAT test. Their Biolog substrate utilization patterns were similar to Pseudomonas syringae pv. syringae’s in Biolog database. Phylogenetic trees with 16S rRNA gene sequences and multilocus sequence typing (MLST) with nucleotide sequences of 4 housekeeping genes, gapA, gltA, gyrB, rpoD and those of P. syringae complex strains in the Plant Associated and Environmental Microbes Database (PAMDB) showed that the green pumpkin isolates formed in the same clade with P. syringae pv. syringae strains. The clade in MLST tree was in the genomospecies 1 group. The phenotypic and genotypic characteristics suggested that the isolates from green pumpkin lesion were P. syringae pv. syringae.
서론
박과 작물은 전 세계적으로 재배되고 있는 채소 작물로서 그 중 애호박을 포함한 호박은 국내에서 약 34만 5천 톤이 생산되고 있다. 재배면적은 1991년 약 5,774 ha였으나 2000년대 들어와서 점차 면적이 확대되어 2014년 재배면적은 9,659 ha로 확인되었다(Ministry of Agriculture, Food and Rural Affairs, 2015). 호박은 덩굴성 1년생 초본식물로 동양계, 서양계, 폐포계로 구분된다. 국내에서 많이 재배되고 있는 애호박과 풋호박은 동양계, 쥬키니호박은 폐포계이며, 밤호박은 서양계로 분류된다. 호박은 과육이 유연하고 비타민 및 무기질이 풍부하여 최근에는 건강 웰빙 식품으로 각광 받고 있으며 음료, 제과 등의 가공식품의 원료로도 널리 이용되고 있다(RDA, 2013).
한국식물병명목록(The Korean Society of Plant Pathology, 2009)에 따르면 국내의 호박에 발생하는 병은 43종으로, 진균에 의한 병 23종, 바이러스에 의한 병 8종, 세균에 의한 병 2종이 보고되어 있다. 호박에 발생하는 세균병은 Pseudomonas syringae pv. lachrymans (Psl)에 의한 세균모무늬병과 Pectobacterium carotovorum subsp. carotovorum에 의한 무름병이다. 2013년 충남 공주의 육묘장과 충북 청원 및 강원 홍천의 애호박 포장에서 기존에서 보고된 병과 다른 새로운 병이 발생하였다. 애호박에서 발생한 병은 호박에 발생이 보고된 세균모늬병과 무름병과는 차이가 있었으며, 본 논문의 첫 번째 저자에 의해 이 병의 발생을 한국식물병리학회에 초록으로 보고하였다(Park 등, 2013). 본 연구에서는 이 병을 일으키는 병원균의 병원성 및 표현형적 · 유전적 특성을 자세하게 조사하고 동정하였다.
재료 및 방법
병원균 분리
충남 공주에서 병징을 가진 애호박를 채집하여 Schaad 등(2001)의 방법에 따라 병원균을 분리하였다. 병징 부위와 건전 부위의 경계 부위를 잘라내어 1% sodium hypochlorite에 3분간 표면 살균하였으며, 멸균수로 2회 세척한 다음 5분간 클린벤치에서 건조하였다. 이 조각을 King’s B 배지(peptone 20 g, magnesium sulphate heptahydrate 1.5 g, dipotassium phosphate 1.5 g, glycerol 15 ml, agar 15 g/l)에 올려놓고 28°C의 배양기에서 2일 배양하여 병반으로부터 자라나온 병원균을 순수배양하였다. 단일 콜로니에서 순수 분리된 병원균은 King’s B 배지에서 배양한 후 20% 글리세롤에 현탁하여 –80°C 냉동고에 동결 보존한 후 실험에 사용하였다.
분리균의 병원성 확인
애호박 병징에서 분리된 NWB SC125, NWB SC127, NWB SC131의 3개 균주를 박과 10개 작물, 애호박(Cucurbita mochata cv. Nongwooae), 풋호박(Cucurbita mochata cv. Bokjuput), 흑종호박(Cucurbita ficifolia), 참박(Lagenaria leucantha cv. Sunbongzhang), 쥬키니호박(Cucurbita pepo cv. Teayang), 오이(Cucumis sativus cv. Seolback), 참외(Cucumis melo var. makuwa cv. Obokggul), 멜론(Cucumis melo cv. Earthtalent), 수박(Citrullus vulgaris cv. Speedggul), 밤호박(Cucurbita maxima cv. Danbam)에 접종하여 병원성을 확인하였다. King’s B 배지에서 28°C 48시간 배양한 세균을 분광광도계 흡광 파장 OD600nm에서 0.1 (약 5×107 cells/ml)로 현탁하였다. 이 현탁액을 50구 트레이에 파종 3주된 1–2 엽기의 10개의 박과 작물에 분무접종하였다. 균주 현탁액 250 ml에 전착제(siloxane) 100 µl를 첨가하였다. 접종 후 18시간 동안 80% 습도의 암조건에 둔 후, 25°C±30°C, 40%–50% 습도의 온실로 옮겨 관리하였다. 접종 7일 후 발병도를 조사하였으며, 발병도 조사는 병발병의 잎면적에 따라 발병수준을 0, 1, 3, 5, 7까지 나누었으며 0은 무발병, 1은 1%–10% 발병면적, 3은 11%–25% 발병면적, 5는 26%–50% 발병면적, 7은 50% 이상의 발병면적으로 정하였다. 발병도(%)는 각 접종구의 발병잎의 면적지수의 총합을 각 접종구의 총조사 잎수의 총 면적지수의 백분율로 구하였다. 본 연구에 한국농업미생물은행(Korean Agriculture Culture Collection, KACC)에서 분양을 받은 P. syringae pv. syringae (Pss) KACC17521과 KACC18392 두 균주를 대조균주로 사용하였다.
전자현미경 관찰
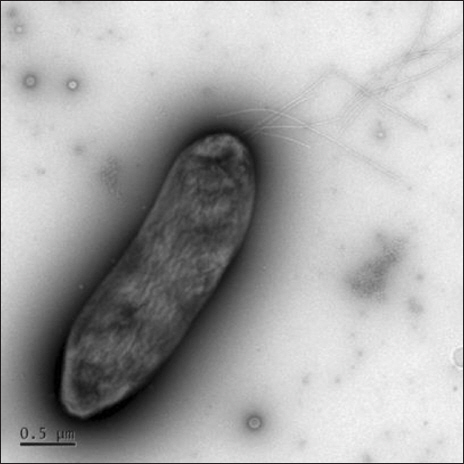
애호박 병징에서 분리한 NWB SC127균주를 투과전자현미경(transmission electron microscopy, Tecnai G2 Spirit; FEI, Hillsboro, OR, USA)으로 관찰하였다. 시료 처리는 200 mash carbon coated grid에 시료를 올리고 2% uranyl acetate를 이용하여 염색한 후 11,000배 배율로 관찰하였다.
형광성, 그람 반응 및 생장온도 조사
분리된 균들을 King’s B 배지에 도말하여 28°C, 3일간 배양한 후 UV350nm 파장에서 형광발생을 확인하였다. 생육온도에 대한 민감도를 확인하기 위하여 King’s B broth 10 ml에 24시간 배양을 한 후 King’s B broth 10 ml에 배양액 100 µl를 접종하여 48시간 동안 진탕배양기(200 rpm)에서 28°C와 35°C 조건으로 배양하였다. 배양 후 균주 현탁액을 King’s B 배지에 도말하여 28°C에 3일간 배양 후 콜로니 형성을 조사하여 생육온도를 조사하였다. Schaad 등(2001)의 방법으로 그람 염색을 하여 현미경으로 관찰하였다.
LOPAT 검정
분리된 병원균 NWB SC127 균주의 LOPAT (levan test, oxidase test, potato soft rot test, arginine dihydrolase activity, tobacco hypersensitive reaction)를 조사하였다(Lelliott과 Stead, 1987; Schaad 등, 2001). Levan 형성을 확인하기 위하여 sucrose peptone agar (5% sucrose nutrient agar) 배지에서 28°C 4일간 배양 후 dome 모양의 콜로니를 형성하는지 관찰하였다. Oxidase 활성은 여과지에 1% 검사용 시약(nitrogen tetramethyl-p-phenolin-diamine dihydrochloride, Sigma)을 적신 후 King’s B 배지에서 24시간 배양된 세균을 시약으로 적신 여과지에 묻힌 후 10초 안에 보라색 또는 청색으로 변하는지 관찰하였다. 감자 무름 반응을 조사하기 위하여 세척한 감자를 화염 멸균한 칼로 0.5 cm 두께로 썰어 절편을 준비하였다. King’s B 배지에 24시간 배양한 병원균을 백금이로 떠서 감자의 절단면에 접종하고, 28°C에서 24–48시간 동안에 접종한 감자에서 무름이 발생하는지 관찰하였다. Arginine dihydrolase 활성을 확인하기 위하여 Thornley’s medium (0.2 g peptone, 1 g NaCl, 0.06 g K2HPO4, 0.6 g agar, 0.02 mg phenol red, 2 g arginine HCl per 200 ml, pH 7.2)을 10 ml씩 시험관에 조제하여 24시간 King’s B 배지에 배양된 균주를 접종하였다. 접종 후 멸균된 미네랄 오일로 시험관 입구를 밀봉한 후 28°C에 배양하여 4일 후 색의 변화를 관찰하였다. 담배에서 과민성 반응은 파종 후 5주된 담배에 King’s B 배지에 24시간 배양한 분리균의 현탁액(약 5×107 cells/ml)을 담배 잎에 주입하여 과민성 반응 여부를 확인하였다.
Biolog 기질 이용성 조사
애호박에서 분리한 NWB SC127과 Pss KACC18392, Psl LMG5172, Pst DC3000 (P. syringae pv. tomato DC3000), Psp KACC10575 (P. syringae pv. pisi KACC10575) 균주를 Biolog GN2 Microplate (Biolog Inc., Hayward, CA, USA)를 이용하여 Biolog 기질 이용성을 조사하였다. 각 균주를 BUG (Biolog Universal Growth) agar에서 30°C 24시간 배양하였다. 배양된 균주를 GN/GP inoculating fluid 용액에 현탁하여 분광광도계 OD600nm에서 52% T 농도가 되도록 희석하였다. 이 현탁액을 Biolog Plate의 각 well에 150 µl씩 분주하여 30°C에서 24시간 배양한 후 변색 여부를 확인하고, Biolog database의 P. syringae pv. syringae data와 비교하였다.
16S rRNA 염기서열 분석
애호박의 병반에서 분리된 7개 균주를 King’s B 배지에 28°C, 24시간 배양하여 (주)코스모진텍(Cosmogenetech, Seoul, Korea)에 16S rRNA sequencing을 의뢰하였다. National Center for Biotechnology Information (NCBI) GenBank에 등록되어 있는 P. syringae complex 균주들의 16S rRNA 염기서열을 이용하여 CLUSTAL W 프로그램으로 염기서열을 다중정렬한 후 분석하였다(Thompson 등, 1994). 유전자 염기서열의 상동성은 Mega (version 5.05; Tamura 등, 2011) BioEdit (version 7.01) 프로그램을 통하여 분석하였다. 정렬된 각 염기서열의 evolutionary distance matrix는 Kimura 2-parameter (K2P)로 구하였고 phylogenetic tree algorithm은 maximum likelihood (ML)를 이용하여 계통도를 그렸으며, 지지도를 확인하기 위하여 1,000번의 bootstrap을 수행하여 500 이상의 값을 표기하였다.
Multilocus sequence typing (MLST) 분석
MLST에 의한 분리균의 계통수(phylogenetic analysis) 분석은 Hwang 등(2005)에 의해 개발된 프라이머를 이용하였다. MLST 분석에 사용된 유전자 염기서열은 총 1,800 bp로 4개의 항존유전자(housekeeping gene) gapA, rpoD, gltA 및 gyrB의 400 bp, 600 bp, 400 bp, 400 bp이다. 본 연구에서 사용된 각 균주들의 염기서열은 Plant Associated and Environmental Microbes Database (PAMDB, http://genome.ppws.vt.edu/cgi-bin/MLST/home.pl)에 등록되어 있는 P. syringae complex 64균주의 해당 항존유전자의 염기서열을 바탕으로 CLUSTAL W 프로그램을 이용하여 다중정렬한 후 분석하였다(Thompson 등, 1994). 유전자 염기서열의 상동성은 Mega (version 5.05) BioEdit (version 7.01) 프로그램을 통하여 분석하였다. 정렬된 각 염기서열의 evolutionary distance matrix는 K2P로 구하였고 phylogenetic tree algorithm은 ML을 이용하여 계통도를 그렸으며, 지지도를 확인하기 위하여 1,000번의 bootstrap을 수행하여 500 이상의 값을 표기하였다.
결과
애호박에 나타난 병징과 분리균의 병원성
충남 공주의 육묘장과 충북 청원 및 강원 홍천의 재배지에서 애호박에 발생한 병의 병징은 초기 유묘의 떡잎 가장자리에 부정형의 강한 수침상이 나타나고(Fig. 1A), 잎에서는 병반 주위에 노란색의 둘레무리(halo)를 가진 회색–갈색의 점무늬가 나타났다(Fig. 1B). 꽃에서는 갈색 수침상의 부정형 병반이(Fig. 1C), 과실에서는 갈색의 움푹 들어간 병반이 나타났다(Fig. 1D).

Symptoms of new disease that occurred on the green pumpkin. Water soaking lesions on leaf (A), spots with strong yellow halo on leaf (B), brown lesion on flower (C), and brown and yellow pit on fruit (D) were observed. Arrows point the lesions.
충남 공주 육묘장의 애호박 병반으로부터 분리된 7개의 균(NWB SC125, NWB SC126, NWB SC127, NWB SC128, NWB SC129, NWB SC130, NWB SC131)은 접종한 유묘의 애호박에서 부정형의 점무늬 병징을 일으켰다. 애호박에서 병원성이 확인된 분리균들 중 NWB SC125, NWB SC127, NWB SC131의 3개 균들과 KACC에서 분양 받은 Pss KACC17521과 Pss KACC18392 두 개 균을 박과 10개 작물 흑종호박, 밤호박, 애호박, 쥬키니호박, 풋호박, 참박, 수박, 참외, 멜론, 오이에 접종하여 병원성을 확인하였다. 그 결과 분양 받은 Pss KACC17521과 Pss KACC18392는 접종한 박과 작물에 병을 일으키지 못하였다. 이 두 균주는 각각 산딸기와 복숭아에서 분리된 균으로 박과 작물에는 병원성이 없는 것으로 판단된다. 분리균 NWB SC125, NWB SC127, NWB SC131은 애호박에 약 70%, 풋호박에 40%, 흑종호박에 60%, 참박에 40%, 쥬키니호박에서는 65%의 발병도를 보였으며, 오이와 참외는 약 10%, 멜론은 5% 이하의 발병도를 보였다. 수박과 밤호박에서는 병을 일으키지 못했다(Table 1, Fig. 2). 접종한 3개의 균주는 기주에 따른 발병도의 차이는 없었다(Table 2).

Disease severity on the 10 Cucurbitaceae plants by inoculation of the three green pumpkin isolates from Gongju-si and Pseudomonas syringae pv. syringae strains from the different host plants

Symptoms on the 10 Cucurbitaceae plants at 7 days after inoculation of the NWB SC127 isolate in green house.
분리균의 형태적·생리적 특성
분리균 NWB SC127을 투과전자현미경으로 관찰한 결과 막대형으로 극에 4개의 편모를 가진 속모균이었다(Fig. 3). NWB SC125, NWB SC127, NWB SC131 균주는 King’s B 배지에서 녹색형광을 보여주었고, 28°C 온도에서는 생육하였으나, 35°C의 온도에서는 생육하지 못하였으며, 그람 반응은 모두 음성으로 나타났다(Table 2). 3가지 분리균은 levan를 형성하였고(양성), oxidase와 arginine dihydrolase의 활성은 음성이었다. 감자 무름 반응은 음성이었고 담배 과민성 반응은 양성으로 5개의 LOPAT group 중 group 1a로 확인되었다(Table 2).
애호박에서 분리한 균주 NWB SC127, Pss KACC18392, Psl LMG5172, Pst DC3000, Psp KACC10575 균주의 Biolog GN2 기질이용성을 조사한 결과, 분리균주 NWB SC127 균주와 Pss KACC18392는 Pss Biolog database와 91%의 동일한 기질이용성을 확인하였다. Pss Biolog database와 다른 P. syringae 균주들과 비교한 결과, Psl LMG5172는 86%, Pst DC3000은 73%, 그리고 Psp KACC10575와 81%의 동일한 기질이용성이 확인되었다. 분리균주 NWB SC127과 Pss KACC18392 두 균주 간에는 96%의 동일한 기질이용성이 확인되었다. 특히, L-Histidine과 D,L-carnitine 기질은 Pss 균들만 양성 반응을 보였다(Table 3, Supplementary Table 1).
분리균의 유전적 특성
NWB SC125, NWB SC127, NWB SC131 균주들의 16S rRNA 염기서열을 얻어 NCBI GenBank에 등록된 P. syringae complex 균들의 16S rRNA 염기서열과 비교하여 계통수를 분석하였다. 분리한 3개 균주들은 모두 동일한 clade로 그룹화되었고, 비교한 P. syringae complex의 균주 중에서 Pss IPPBC-R30과 같은 clade로 그룹화되었다(Fig. 4). 항존유전자 4개 유전자의 염기서열을 이용한 MLST 분석에서는 NWB SC125, NWB SC127, NWB SC131 균주들은 독립된 clade를 형성하였고, P. syringae Cit7, Pss 1212R, Pss 61과 가장 유사한 그룹으로 확인되었다(Fig. 5). 이 clade는 P. syringae pv. pisi, P. syringae pv. atrofaciens, P. syringae pv. japonica 균주들을 포함하고 있는 genomospecies group 1에 속하였다(Fig. 5). 오이 및 호박에서 세균모무늬병을 일으키는 Psl은 genomospecies group 2에 속해 애호박 분리균과는 다른 계통군이었다(Fig. 5).

Maximum likelihood tree inferred from 16S rRNA gene sequences of the green pumpkin isolates (NWB SC125, 127, 131) and Pseudomonas syringae strains. Bootstrap values (≥50%) based on 1,000 replicates are shown at the branches. The bar indicates the nucleotide substitutions per site.

Maximum likelihood tree inferred from gapA, rpoD, gltA, gyrB sequences of the green pumpkin isolates (NWB SC125, 127, 131) and Pseudomonas and other bacteria. The 4 gene sequences of Pseudomonas and other bacteria were obtained from the Plant Associated and Environmental Microbes Database (PAMDB, http://genome.ppws.vt.edu/cgi-bin/MLST/home.pl). Bootstrap values (≥70%) based on 1,000 replicates are shown at the branches. The bar indicates the nucleotide substitutions per site. Gsp is genomospecies.
고찰
본 연구에서는 2013년 충남 공주의 육묘장과 국내의 일부 재배지에서 새롭게 발생한 애호박 점무늬병 증상에서 병원균을 분리하고, 분리된 균들의 병원성을 확인하였으며, 표현적 유전적 특성을 분석하여 동정하였다. 애호박에서 발생한 병징은 강한 수침상, 점무늬 주위의 노란색 둘레무리 등의 전형적인 세균에 의한 병의 특징을 보였으며, 병반에서 진균은 관찰되지 않았다. 따라서 병징은 세균에 의한 점무늬병 병징으로 추정되었으며, 이 병징의 특징들은 국내에 보고된 2종의 호박 세균병인 세균모무늬병과 무름병의 병징과는 차이가 있었다(The Korean Society of Plant Pathology, 2009). 이 병반에서 분리된 분리균은 박과 10개 작물 중에서 애호박, 쥬키니호박, 풋호박, 흑종호박, 참박, 참외, 멜론, 오이에서 병원성이 확인되었고, 수박과 밤호박에서는 병반을 형성하지 않았다. 분리균은 밤호박을 제외한 호박류에서 높은 발병도를 보였다. 애호박, 풋호박은 동양계(Cucurbit moschata)이고, 쥬키니호박은 폐포계(Cucurbit pepo)이며 흑종호박(Cucurbita ficifolia) 하나의 종으로 분류되고 있으며, 분리균이 병반을 형성하지 않은 밤호박은 서양계(Cucurbit maxima)였다(RDA, 2013). 따라서 애호박 분리균은 호박의 계통에 따라 병원성이 차이가 나는 것으로 생각된다.
호박류에서 병원성이 확인된 분리균주는 King’s B 배지에서 형광을 보이는 그람음성의 균이었고, 막대형의 4개의 편모를 가진 속모균이었다. 또한 35°C 이상의 온도에서는 생육이 불가능하였으며, Lelliott 등(1966)이 제안한 LOPAT test를 통하여 LOPAT group 중 group 1a에 속하는 것을 확인하였다. 이상의 결과는 분리균이 P. syringae의 특성을 가졌음을 의미한다. 16S rRNA 염기서열과 NCBI GenBank database에 등록되어 있는 다른 P. syringae complex의 16S rRNA 염기서열을 이용한 계통도에서 애호박 분리균들은 P. syringae pv. syringae (Pss)의 균주와 같은 그룹(clade)으로 분류되었지만, 일부 다른 Pss는 다른 그룹(clade)으로 그룹화되었다. 16S rRNA 유전자 염기서열은 균주 사이의 유사도가 매우 높아 P. syringae complex 내의 pathovar (pv.)의 분류 기준으로 적합하지 않는 것으로 알려져 있다(Young, 2010). Biolog의 기질 이용성을 다른 P. syringae 균주들과 비교한 결과 애호박 분리균은 Biolog의 database에 등록되어있는 Pss 균주의 기질 이용성과 91% 동일한 것으로 나타났다. 특히 Krejzar 등(2004)의 발표 따르면 Pss는 다른 P. syringae 균들과는 다르게 D,L-carnitine 기질이용성에 양성반응을 보인다고 보고하였는데, 본 연구 결과에서 NWB SC127과 Pss KACC18392 균주들만 D,L-carnitine 기질이용성에 양성반응을 보여 분리된 NWB SC127 균주는 P. syringae pv. syringae일 가능성이 높은 것으로 나타났다.
P. syringae complex를 1999년 Gardan 등이 DNA-DNA hybridization을 통한 9개의 genomospecies group으로 분류하였고, Bull 등(2011)이 MLST 분석으로 P. syringae complex의 9개 genomospecies group이 존재함을 다시 확인하였다. 애호박에서 분리한 3가지 균주의 4가지(gapA, rpoD, gltA, gyrB) 항존유전자 염기서열을 이용한 계통수는 이 균주들이 Pss와 가장 유사한 clade에 속하며, genomospecies group 1에 속한 것으로 보여주었다. 박과에서 세균모무늬병을 일으키는 P. syringae pv. lachrymans는 genomospecies group 2에 속한다. 본 연구의 결과로 확인된 애호박 분리균의 형태적, 생리생화학적, 유전적 특성을 바탕으로 이들 분리균을 P. syringae pv. syringae로 판단하였다.
국내에서는 Pss가 호박에 병을 일으키는 것이 아직까지 보고되지 않았지만 국외에서는 여러 번 있었다. 미국의 조지아 주에서 폐포계 호박에서 Pss에 의한 잎점무늬병의 발생이 보고되었다(Langston Jr 등, 2003). 세르비아의 폐포계 호박(oil pumpkin)에서 Pss에 의해 잎에 점무늬병이 발생하여 피해를 주었고(Balaž 등, 2014), 최근에는 미국 플로리다, 조지아, 캘리포니아 주에서 수박, 멜론, 호박의 병든 잎에서 27개의 P. syringae strain을 분리하였고, MLST와 BOX-PCR 분석으로 27개의 균들 중 26개의 균이 genomospecies group 1에 속한다고 보고하였다(Newberry 등, 2016). 따라서 Pss에 의해 호박에서 병이 발생하는 것은 전 세계적으로 이번이 처음은 아니다.
본 연구를 통해 애호박의 점무늬병징에서 분리한 균주들은 동양계 호박과 폐포계 호박, 흑종호박에 병원성을 가지고 있었으며, P. syringae pv. syringae로 동정되었다. 현재까지 국내에서 애호박에 P. syringae pv. syringae에 의한 병명은 등록되어 있지 않다. 따라서 애호박에서 새롭게 발생한 이 병을 “애호박 세균점무늬병”으로 제안한다.
요약
육묘장과 재배포장의 애호박에서 새롭게 발생한 병원균의 특성을 분석하여 동정하였다. 병징은 잎에 수침상의 병반과 강한 노란색의 둘레무리를 가진 점무늬, 꽃에서 갈색병반, 열매에 노란색의 점무늬였다. 잎의 점무늬로부터 분리된 세균은 박과작물 8개의 식물체, 흑종호박, 애호박, 쥬키니호박, 풋호박, 참박, 참외, 멜론, 오이에 병원성이 있었지만 수박과 밤호박에서는 병을 일으키지 못했다. 분리세균은 다발모를 가진 막대형으로 그람음성이며 King’s B 배지에서 형광성이었으며, LOPAT 1a 그룹에 속했다. 그들의 Biolog 기질이용성은 Biolog database의 P. syringae pv. syringae 이용성과 유사하였다. 16S rRNA 유전자 염기서열을 이용한 계통수와 4개의 항존유전자(gapA, gltA, gyrB, rpoD)의 염기서열과 PAMDB에 있는 P. syringae 균주들의 염기서열을 이용한 MLST는 애호박 분리균주들이 P. syringae pv. syringae 균주들과 동일한 그룹(clade)으로 그룹화되었고, MLST 계통수에서 애호박 분리균주들의 그룹(clade)은 genomospecies 1에 속하였다. 표현형적, 유전적 특성은 애호박 분리균이 P. syringae pv. syringae임을 제시하였다.
Conflicts of Interest
No potential conflict of interest relevant to this article was reported.