벼 도열병 단일 저항성 유전자를 이용한 도열병균의 병원형 분류
Pathotype Classification of Korean Rice Blast Isolates Using Monogenic Lines for Rice Blast Resistance
Article information
Abstract
The rice blast fungus is a representative model phytopathogenic fungus in which Gene-for-Gene interaction with host rice is applicable. After 1980, eight differential varieties have been constructed and classified to analyze the race of rice blast isolates in Korea. However, since there is limited information about the genetic background of rice blast resistance genes within the Korean differentials, scientific analysis on the emergence of new race or resistance break down was difficult. Recently, a differential system has been developed using monogenic resistance lines to understand the interactions of pathogen race and rice resistance genes. In this study, a total of 50 isolates were selected from four different races isolated in Korea, and they were inoculated into monogenic lines. As a result, the isolates in the same race classified by the Korean differential system reacted differently in single monogenic lines. This suggests that the isolates categorized as the same race group contains different avirulence genes and furthermore, it is presumed that the Korean differential system is difficult to provide useful information for breeding program. For this reason, introduction of differential system using monogenic resistance lines is required in addition to the current system.
서론
벼 도열병균 Magnaporthe oryzae는 벼의 전 생육기에 병을 일으키며, 특히 수확기에 환경조건이 벼 도열병균에 유리할 경우 엄청난 수량 손실을 초래한다. 벼 도열병 감수성 품종의 경우 벼 생산량의 65%까지의 수확량 감소가 발생한다고 보고된 바 있다(Li 등, 2007).
특히 벼 도열병균은 기주인 벼와 Gene-for-Gene 상호작용이 적용 가능한 대표적인 모델 식물병원성 곰팡이다. 현재까지 기주인 벼의 저항성(Resistance) 유전자는 40개 이상이 밝혀졌으며, 이에 상응하는 병원균의 비병원성(Avirulence) 역시 9개의 유전자가 분자생물학적 그리고 기능유전학적으로 밝혀졌다(Bohnert 등, 2004; Farman 등, 2002; Jia 등, 2000; Kang 등, 1995; Li 등, 2009; Orbach 등, 2000). 벼 도열병균 방제 전략으로 육종을 통한 저항성 유전자 도입이 가장 효과적으로 생각되고 있다. 따라서 집단 수준에서 벼 도열병균의 비병원성 유전자의 분포 연구는 레이스(race) 혹은 병원형(pathotype)을 이해할 수 있을 뿐만 아니라, 벼 품종 내 저항성 유전자를 도입하기 위한 유용한 정보를 제공할 수 있을 것이다.
우리나라는 1962년부터 벼 도열병균의 레이스 분화를 이해하기 위해 연구가 시작된 이래로(Ahn과 Jung, 1962), 지금까지 레이스 검정이 이루어지고 있다. 특히, 현재 이용되고 있는 한국형 판별 품종은 1980년도 Lee 등에 의해 고안되었는데, 이 방법은 인디카(Indica) 및 통일계 품종 4개(cvs. Tetep, 태백 (Taebaeg), 통일(Tongil), 유신(Yusin))와 자포니카(Japonica) 계열 품종 4개(cvs. Kanto51, 농백(Nongbaeg), 진흥(Jinheung)과 낙동(Nagdong))로 구성되어 있다(Lee 등, 1987). 이 8개의 판별 품종에서 나타나는 균주의 친화적(compatible) 혹은 비친화적 (incompatible) 반응을 통해 레이스를 검정하는 방법이다(Lee 등, 1987). 그러나, 이 한국형 판별 품종 중 인디카 및 통일계 품종은 현재 우리나라에서는 재배되는 주요 품종이 아니며, 한국형 판별 품종은 단인자 형태의 저항성 유전자형이 아니기 때문에 도열병균의 비병원성 유전자의 유무, 새로운 레이스의 출현 및 병 저항성 붕괴를 과학적으로 분석하기 어려운 점이 있다. 실제로 한국형 판별 품종을 통해 구분된 레이스와 분자생물학적 마커를 통한 레이스-DNA fingerprinting 연구 간의 연관성을 밝히는 연구들이 시도되었으나(George 등 1998; Park 등, 2008), 한국형 판병 품종을 통한 레이스와 transposon을 이용한 분자생물학적 마커는 상관관계가 없는 것으로 나타났다.
이러한 한국형 판별품종을 통한 레이스 분화 연구는 최근까지 연구되어 왔는데, 그 결과에 따르면 우리나라 전역에 벼 도열병이 감소하고 있음에도 불구하고 병원형 레이스는 과거에 비해 더욱더 다양해지고 있는 것으로 나타났다(Kim 등, 2016). 이러한 다양한 레이스의 분화는 벼 도열병균의 유전적 변이에 의한 것으로 추정되며, 나아가 저항성 품종에 대한 친화성 균주 집단이 증가될 가능성으로 이어질 수 있을 것으로 생각되고 있다(Han 등, 2001). 이러한 저항성 품종이 감수성 품종으로 바뀌는 저항성 역전 현상은 이미 벼를 비롯한 다수의 작물에서 보고된 바 있다(Mundt, 2014).
Hayashi와 Fukuta (2009)는 벼 도열병균의 비병원성 유전자 의 유무를 알아보기 위해 국제미작연구소(International Rice Research Institute, IRRI)에서 개발된 monogenic lines(단인자 계통)을 이용한 새로운 판별시스템을 제안하였다. 이 판별 시스템은 벼 도열병에 대항하는 23개 저항성 유전자(Pish, Pib, Pit, Pia, Pii, Pi3(t), Pi5(t), Pik-s, Pik-m, Pi1, Pik-h, Pik, Pik-p, Pi7(t), Pi9, Piz, Piz-5, Piz-t, Pita-2, Pi12(t), Pita, Pi19(t), Pi20(t))를 벼 품종 cv. LTH에 각각 넣어 단인자 저항성 유전자를 갖고있는 시스템이다. Hayashi와 Fukuta (2009)는 이들 monogenic lines 각각을 유전자좌에 따라 5개의 그룹으로 나누고, 그에 해당하는 monogenic lines에 병원균의 병원성 반응을 코드화하여 5개 의 숫자조합으로 도열병균을 분류하였다. 이 시스템으로 분류할 경우 저항성 유전자에 대한 벼 도열병균이 가지고 있는 비병원성 유전자를 직관적으로 추정할 수 있으며, 비병원성 유전자 유무를 통해 병원형을 분류할 수 있을 것이라고 제안하였다 (Hayashi와 Fukuta, 2009).
본 연구에는 한국형 판별시스템에 의해 분류된 벼 도열병균의 대표균주를 선발하고, 선발된 대표 균주를 LTH 벼 품종에 기반한 monogenic line에 접종하여 병원성 검정을 하였다. 또한, 병원성 검정 결과를 Hayashi와 Fukuta (2009)가 제안한 방법을 적용하여 분류해 보았을 때, 비병원성 유전자를 추정할 수 있는지를 알아보았다. 본 연구 결과는 향후 새로운 한국형 판별 시스템 구축에 기초 자료로 이용될 수 있을 것이며, 연구 결과로 밝혀진 비병원성 유전자의 분포는 벼 도열병 저항성 품종 육종에 기초 자료로 사용될 수 있을 것이다.
재료 및 방법
벼 도열병 균주
벼 도열병균은 국립식량과학원 재배환경 과에 보관된 50개 균주를 선발하여 실험에 사용하였다. 선발 기준으로 지역별, 품종별, 그리고 수집 연도를 고려하여 선발하였으며, 한국 판별 품종 체계에서 판별된 레이스들 중 KI101, KI201, KI401 및 KJ101에 해당하는 균주를 선발하여 실험에 사용하였다.
품종 및 계통
실험에 사용한 monogenic line (단인자 계통)은 국제미작연구소에서 중국 자포니카 이병성 품종 LTH에 단일 저항성 유전자 23개(Pish, Pib, Pit, Pia, Pii, Pi3(t), Pi5(t), Piks, Pik-m, Pi1, Pik-h, Pik, Pik-p, Pi7(t), Pi9, Piz, Piz-5, Piz-t, Pita-2, Pi12(t), Pita, Pi19(t), Pi20(t))를 도입하여 육성한 26계통(IRBLsh-S, IRBLb-B, IRBLt-K59, IRBLa-A, IRBLi-F5, IRBL3-CP4, IRBL5-M, IRBLks-S, IRBLkm-Ts, IRBL1-CL, IRBLKh-K3, IRBLk-Ka, IRBLkp-K60, IRBL7-M, IRBL9-W, IRBLz-Fu, IRBLz5-CA, IRBLzt-T, IRBLta2-Pi, IRBLta2-Re, IRBL12-M, IRBLta-K1, IRBLta-CP1, IRBL19-A, IRBL20-IR24) 및 대조군으로 LTH를 병원성 실험에 사용하였다 (Tsunematsu 등, 2000).
단인자 벼 육묘
대조군 LTH와 26종의 monogenic lines에 대한 벼 도열병균의 병원성을 검정하기 위해 각각의 벼 종자는 1% 클로락스를 이용하여 30분간 표면 살균하여 3-6일간 발아시켰다. 그 후 4×4×4 cm3 플라스틱 포트에 각각 품종당 5립씩 발아된 벼를 심었다. 파종한 토양은 벼 못자리용 상토(질소전량 800-2,500 mg/kg, 유효인산 150-650 mg/l, EC 2.0 dS/m 이하)를 사용하였으며, 다른 미생물에 의한 오염을 차단하기 위해 격리된 온실에서 3-4엽기까지 키웠다.
접종원 준비
접종원 준비를 위해 쌀겨배지(rice polish agar: 쌀겨 20 g, 설탕 20 g, 한천 20 g, 증류수 1 l)를 40 ml씩 페트리-디쉬에 분주하여 굳힌 다음, 감자한천배지(potato dextrose agar)에서 10일 자란 균총 절편을 2 ml의 살균 증류수에 넣었다. 살균된 봉을 이용하여 균사절편을 마쇄 후, 마쇄한 현탁액을 쌀겨배지에 접종하였다. 접종한 페트리-디쉬는 26°C 항온기 에서 균사가 쌀겨배지 표면에 가득 자랄 때까지 7일간 배양하였다. 멸균된 고부 브러쉬로 배양된 쌀겨배지에서 만들어진 기중균사를 제거하고 페트리-디쉬의 뚜껑을 열어 25°C의 지속적인 광조건 배양기에서 3일간 배양하여 분생포자를 유도하였다. 포자 수거를 위해 분생포자가 형성된 쌀겨배지에 0.2% Tween 20 용액을 10 ml을 넣어 멸균된 고무 브러시를 이용하여 분생 포자를 수거한 뒤 거즈로 걸러 균사를 제거하였다. 분생포자 현탁액은 2×105/ml의 포자 농도로 맞춘 뒤, 총 25 ml의 포자 현탁액을 실험에 사용하였다.
병원성 검정
3-4엽기의 대조군 LTH와 26종의 monogenic lines에 준비된 포자 현탁액을 50 ml 시험관에 넣어 특별 제작 한 유리 스프레이를 이용하여 안개처럼 작은 물 입자로 분사되도록 진공 콤프레샤를 이용하여 25 ml씩 벼 잎에 골고루 분무 되도록 접종하였다. 접종된 벼는 26°C 포화 습도 접종상에 넣어 24시간 동안 암 조건에서 습실 처리 한 후, 온실에 옮겨 7 일간 배양하였다. 발병 조사는 식물체 전체 잎에 형성된 병반형 및 병반수를 계수하여 국제미작연구소의 조사 기준(IRRI, 1988)에 따라 발병 지수 0-5로 조사하였다. 0-2는 저항성, 3은 중도이병성, 4-5는 이병성으로 구분하여 단계별로 조사하였으며, 최종적으로 0-3은 저항성(R)으로, 4-5는 이병성(S)으로 판정하였다.
결과 및 고찰
한국형 판별시스템으로 분류된 벼 도열병 균 선별
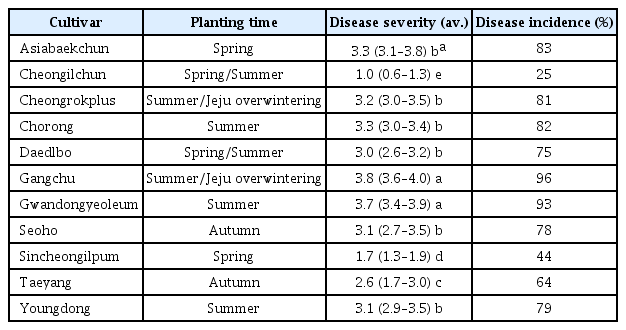
한국형 판별시스템은 8개의 선별된 품종에 대한 병원성 반응에 따라 KI와 KJ 레이스로 구분하고 있다(Lee 등, 1987). 판별 품종 모두에 감수성을 보일 경우 KI101 레이스로 판정하며, Tetep에서만 감수성 반응을 보일 경우 KI201 레이스, Tetep을 비롯하여 태백과 통일벼에 저항성을 보이는 경우 KI401 레이스로 판정하며, Tetep과 3개의 통일계 품종인 태백, 통일 그리고 유신벼에 저항성을 보일 경우 KJ101 레이스로 판정한다(Table 1). 본 실험에서는 KI101, KI201, KJ101 및 KI401에 속한 균주들을 각각 6개, 6개, 11개 및 27개 선발하여 실험에 사용하였다. 각 선발된 균주는 Table 1에서 보여준 8개의 벼 품종에 대한 병 반응이 레이스 별로 모두 동일하게 나타났다.
단인자 저항성 계통에 대한 우리나라 균주의 병원성 검정 결과
선발된 50개의 벼 도열병균을 LTH품종에 기반하는 monogenic lines에 병원성을 검정하였다. 검정 결과는 실험을 수행한 총 50개 중 KI101 (15-14 및 15-16 균주), KI201 (15-40 및 15-161 균주), KI401 (95-68 및 95-259 균주)과 KJ101 (95-58 및 95-69 균주) 레이스 중 2개씩의 균주 결과를 기술하였다(Table 2). 그 결과, 15-14와 15-16은 동일한 KI101 레이스에 속함에도 불구하고 9개의 monogenic lines (t-K59, km-Ts, kh- K3, 9-W, z5-CA, zt-T, ta2-Pi, ta-CP1, 19-A 및 20-IR24)에 대해 서로 다른 저항성과 감수성을 보였다. 또한, 15-40과 15-161의 경우 동일한 KI201 레이스이지만 13개의 monogenic lines (sh-B, t-K59, km-Ts, kh-K3, kp-K60, 9-W, zt-T, ta2-Pi, 12-M, ta-K1, ta-CP1, 19-A 및 20-IR24), 95-68과 95-259의 경우 13개의 monogenic lines (t-K59, km-Ts, kh-K3, kp-K60, 7-M, z-Fu, z5-CA, zt-T, ta2-Pi, ta-K1, ta-CP1, 19-A 및 20-IR24), 그리고 KJ101 레이스 들인 95-58과 95-69 균주의 경우 12개의 monogenic lines (t-K59, i-F5, ks-F5, km-Ts, 1-CL, 9-W, z-Fu, zt-T, ta2-Re, 12-M, ta-CP1 및 20-IR24)에서 서로 다른 병원성 결과를 보였다(Table 2). 이 결과는 실험에 사용한 모든 균주를 기술 할 경우 동일 레이스내의 균주간 불일치가 더욱 복잡하게 나타나는데, 이 결과는 한국의 판별품종을 통해 분류된 레이스가 벼의 저항성(resistance) 유전자와 벼 도열병균의 비병원성(Avirulence) 유전자간의 상호작용을 통한 분류에 적용할 수 없음을 보여준다.
이 연구 결과를 통해 우리는 직관적으로 15-14 균주가 저항성 유전자 Pit, Pikk-m, Pik-h, Pi9, Piz-5, Piz-1, Pita-2, Pita, Pi20에 대항하는 비병원성 유전자를 갖고 있는 것을 유추할 수 있었으며, 15-16 균주의 경우 저항성 유전자 Pi9, Pi19, Pi20에 대항하는 비병원성 유전자를 갖고 있는 것을 추정할 수 있었다. 이러한 정보를 지속적으로 수집한다면, 집단 수준에서 비병원성 유전자 분포를 통한 저항성 벼 육종에 유용한 정보를 제공할 수 있을 것이다. 그러나, 아직까지 밝혀지지 않은 저항성 유전자와 그에 대항하는 비병원성 유전자가 있을 수도 있기에 단정할 수 없지만, 현재 사용되고 있는 한국형 판별 시스템은 벼와 병원균의 Gene-for-Gene 상호작용을 설명하기에는 한계가 있다.
국제 판별 시스템 적용
Hayashi와 Fukuta (2009)는 monogenic lines을 5개 그룹으로 나누어 분류하였는데, 그룹 I (Sh-B, LTH, b-B, a-A 그리고 t-K59), 그룹 II를 저항성 유전자 Pii 유전자좌 기반하여(i-P5, 3-CP4 그리고 5-M), 그룹 III를 Pik 유전자좌에 기반하여(ks-F5, km-Ts, k-Ka, 1-CL, kp-K60, kh-K3 그리고 7-M), 그룹 IV를 Piz 유전자좌에 기반하여(9-W, z-Fu, z5-CA 그리고 zt-T), 그리고 그룹 V를 Pita 유전자좌에 기반하여(ta2-Pi, ta-K1, 19- A, ta2-Re, ta-CP1, 20-IR-24 그리고 12-M)으로 나누었고, 각 그룹에 속한 계통에 대해 Gilmour’s method를 이용해(Gilmour, 1973) 이병성 코드를 부과하여 그룹명과 각 코드를 조합하여 레이스 분류하는 시스템을 개발하였다(Table 3).
이번 연구에서 우리는 선별된 한국 균주의 단인자 계통에 대한 이병성 결과를 JIRCAS에서 제안한 국제 판별시스템에 적용 해 보았다(Table 3). 예를 들어, 15-14 균주의 병원성 테스트 결과 그룹 I에 속한 계통 중 IRBLt-K59를 제외한 모든 계통에 이병성 반응을 보여 분류 명칭 첫 번째 부분은 33으로 구분하였고, 그룹 II에 속한 계통 IRBLi-F5, IRBL3-CP4와 IRBL5-M에 이병성 반응을 보여 분류명칭 두 번째 부분은 그룹명과 병원성 반응 코드를 반영하여 i7으로 구분하였다. 그룹 III의 IRBLkm-Ts 와 IRBLkh-K3에는 저항성 반응을 보였고, IRBLks-F5, IRBL1-CL, IRBLk-Ka, IRBLkp-K60, IRBL7-M에는 이병성 반응을 보여 k127로 명명할 수 있다. 저항성 유전자 Piz의 유전자좌가 속해있는 그룹 IV의 계통중 IRBLz-Fu에는 이병성을, IRBL9-W, IRBLz5-CA와 IRBLzt-T 계통에는 저항성 반응을 보여 z01로 구분하였고, 저항성 유전자 Pita 유전자좌 그룹에 속한 계통중 IRBLta2- Pi, IRBLta-CP1, IRBL20-IR24에는 저항성 반응과 IRBLta2-Re, IRBL12-M, IRBLta-K1, IRBL19-A에는 이병성 반응을 보여 ta611 로 구분하였다.
결과적으로 15-14 균주는 다섯 그룹에 속한 단인자 저항성 계통에 대한 병원성 반응 결과에 따른 분류 체계로 33-i7-k127- z01-ta611로 분류할 수 있었다(Hayashi와 Fukuta, 2009). 같은 방법으로 균주 15-40는 63-i7-k125-z13-ta710으로 분류될 수 있고, 균주 06-85는 13-i3-k151-z02-ta000으로, 95-58은 33-i7- k127-z01-ta611로 분류될 수 있다(Table 3).
한국형 판별 시스템에 따라 KI/KJ레이스로 분류된 50개 균주에 대해 같은 방법으로 명명한 결과를 Table 4에 나타내었다. 그 결과 같은 레이스로 분류된 균주들의 단인자 저항성 계통에 의한 분류가 매우 다양하게 나타났는데, 50개 균주에 대해 49개의 pathotype으로 분류 되었다(Table 4). 이 코드 분류표를 통해 각 병원균에 대한 23개의 벼 도열병 저항성 유전자에 대응하는 반응을 분류 코드로 확인할 수 있었으며, 저항성 유전자에 대응하는 비병원성 유전자의 유무를 추정할 수 있었다. 그러나, Hayashi와 Fukuta (2009)가 제안한 분류 코드는 직관적으로 이해하기 매우 복잡하고 어려운 것으로 판단되었다. 따라서, 좀 더 쉽고 직관적으로 이해할 수 있는 한국형 분류체계 를 앞으로 개발해야 할 것이다.
도열병균은 저항성 Gene-for-Gene 반응이 매우 명확한 병원균이다. 한국형 판별품종이 보유하고 있는 병 저항성 유전자가 불분명하여 지금까지 분류된 레이스 반응을 통해 벼 도열병균의 비병원성 유전자의 분포를 이해하기에 어려웠다. 그러나, 이번 연구를 통해 한국에서 분리된 벼 도열병균이 가지고 있는 비병원성 유전자들의 추정할 수 있었으며, 이러한 정보를 지속적으로 확보할 경우 기후변화에 따른 돌발적으로 발생 가능한 벼 도열병을 저항성 유전자 도입을 통한 육종학적인 방제에 큰 도움을 줄 수 있을 것이다.
요약
벼 도열병은 벼를 재배하는 지역에서는 가장 중요한 병 중 하나이다. 특히, 벼 도열병균은 기주인 벼와 Gene-for-Gene 상호 작용이 적용 가능한 대표적인 모델 식물병원성 곰팡이다. 우리나라는 1980년 이래로 벼 도열병균의 레이스를 분석하기 위해 8개의 판별 품종을 이용한 시스템을 구축하여 분류하였다. 그러나 이 판별 품종이 어떤 저항성 유전자를 가지고 있는지에 관해 명확한 정보가 없어 새로운 레이스의 출현이나 병 저항성 붕괴 등에 대하여 과학적인 분석이 어려웠다. 최근 병원균의 레이스와 벼의 저항성 유전자의 상호작용 이해를 돕기 위해 LTH 품종에 단인자 저항성 계통을 각각 다르게 도입한 판별시스템이 개발되었다. 본 연구에서는 우리나라의 1995년부터 2015년까지 분리된 4개의 다른 레이스 KI101, KI201, KI401 및 KJ101로 부터 총 50개 균주를 선발하여 LTH 품종에 기반한 단인자 저항성 계통에 접종하여 그 결과를 이전 레이스와 비교 분석해 보았다. 그 결과 한국형 판별시스템으로 분류된 동일 레이스내의 균주들이 단인자 계통에서 서로 다른 반응을 보였다. 이 결과 동일 레이스에 속하는 균주들이 서로 다른 비병원성 유전자를 지닌 것을 의미하며, 더 나아가 새로운 저항성 벼 품종 육종에 유용한 정보를 제공하기 어려울 것으로 추정되었다. 이 연구 결과 현재의 판별시스템과 더불어 단인자 저항성 품종을 통한 판별시스템 도입이 요구되었다. 이 연구 결과는 향후 한국의 판별 시스템 개발에 기초 자료로 활용 될 수 있을 것이다.
Acknowledgement
This work was carried out with the support of “Cooperative Research Program for Agriculture Science & Technology Development (Project title: Monitoring of race change of rice blast and analysis of genetic diversity program. Project No. PJ01119401)” Rural Development Administration, Republic of Korea.
References
Notes
Conflicts of Interest
The authors declare that they have no competing and commercial interests in this work.