우리나라에서 분리한 참다래 궤양병균 Pseudomonas syringae pv. actinidiae 균주들의 Biovar
Biovars of Pseudomonas syringae pv. actinidiae Strains, the Causal Agent of Bacterial Canker of Kiwifruit, Isolated in Korea
Article information
Abstract
Pseudomonas syringae pv. actinidiae is the causative agent of bacterial canker of kiwifruit. The population of this pathogen is differentiated into three biovars, biovar 1, 2 and 3, according to their molecular characteristics. In this work, we determined biovars of P. syringae pv. actinidiae strains isolated in Korea since 1997 and stored in Department of Biology, Sunchon National University, Suncheon, Korea. The biovars of P. syringae pv. actinidiae strains were determined by PCR using biovar specific primers developed previously. Of 682 strains investigated, 288 strains belonged to biovar 2, while 394 strains were biovar 3. There were no P. syringae pv. actinidiae strains belonging to biovar 1 among the strains isolated in Korea. Sudden outbreak and spreading of bacterial canker caused by biovar 3 strain suggest that this strain has character of rapid transmission.
서론
Pseudomonas syringae pv. actinidiae는 참다래 궤양병의 원인세균이다. 이 세균은 1984년 일본에서 녹색 과육의 참다래인 Actinidia deliciosa의 궤양병 병징에서 처음 분리되어 P. syringae의 새로운 pathovar인 pv. actinidiae로 명명되었다(Takikawa 등, 1989). 중국에서는 같은 시기에 Hunan성에서 궤양병의 발생이 보고된 이후(Fang 등, 1990), 참다래 주요 재배지역인 Anhui, Sichuan, Shaanxi (Liang 등, 2000; Liu 등, 1996) 등에서 P. syringae pv. actinidiae에 의한 참다래 궤양병의 발생이 보고되었다. 우리나라와 이탈리아에서는 1994년 처음 보고된 바 있다(Koh 등, 1994; Scortichini, 1994). 그러나 뉴질랜드를 비롯한 그 밖의 다른 참다래 생산국에서 궤양병이 보고된 바는 없었다. 일본과 이탈리아에서 분리한 P. syringae pv. actinidiae 집단의 균주들은 유전적으로 동일하였으나 우리나라에서 분리된 균주와는 서로 달랐다(Han 등, 2003). 흥미롭게도 일본과 우리나라에서는 참다래 과수원을 폐원시킬 만큼 심각한 피해를 주었으나 이탈리아에서는 비교적 약한 증상을 보였을 뿐 폐원시킬 정도의 병징은 나타나지 않았다(Ferrante와 Scortichini, 2010).
그러나 2008년 중부 이탈리아에 위치한 과수원에서 황색 과육의 참다래인 Actinidia chinensis로부터 종전과 달리 병징이 심각한 궤양병이 발생하여, A. deliciosa 과수원을 포함한 이탈리아 전체 참다래 과수원으로 빠른 속도로 전파되기 시작하였다(Balestra 등, 2009; Ferrante와 Scortichini, 2009). 이 병원균은 곧이어 다른 유럽 국가인 포르투갈(Balestra 등, 2010), 스페인(Balestra 등, 2011), 프랑스(Vanneste 등, 2011), 스위스(EPPO, 2011), 터키(Bastas와 Karakaya, 2012) 등에서 보고되었고, 계속해서 독일(EPPO, 2013), 슬로베니아(Dreo 등, 2014), 그리스(Holeva 등, 2015) 등으로 전파되었다. 뉴질랜드와 칠레에서도 2010년부터 궤양병이 발견되기 시작하였으며(Everett 등, 2011; ProMED-mail, 2011), 우리나라에서도 같은 유전자형의 균주가 2011년부터 발견되기 시작하였다(Koh 등, 2012). 1980년대부터 우리나라와 일본에서 발견된 균주와는 다른 새로운 유전자형을 갖는 P. syringae pv. actinidiae 균주에 의한 궤양병의 대발생은 전 세계적으로 참다래 산업에 치명적인 타격을 안겨주고 있다.
병원성과 전파성이 강한 새로운 균주의 출현은 P. syringae pv. actinidiae 집단에 대한 유전적 특성을 비교 조사하는 계기가 되었다. Chapman 등(2012)은 7개의 house keeping 유전자와 11개의 effector 유전자의 다좌위 서열형(multi-locus sequence typing, MLST) 분석을 통하여 P. syringae pv. actinidiae 집단을 Psa1–4의 4개의 biovar로 분류하였다. Psa1 (이하 biovar 1으로 칭함)은 일본과 1992년 이탈리아에서 분리된 균주들이었고, biovar 2 (Psa2)는 우리나라에서 분리된 균주들이었다. Biovar 3 (Psa3)는 2008년 이후 이탈리아, 뉴질랜드, 칠레, 중국 등에서 분리된 균주였다. Biovar 4 (Psa4)는 잎에 반점을 일으키는 정도만의 약한 증세를 보이는 균주로 오스트레일리아와 뉴질랜드에서 보고된 균주들이었다. 최근 Biovar 4는 P. syringae의 새로운 pathovar인 pv. actinidifoliorum으로 명명됨으로써 현재 P. syringae pv. actinidiae 집단은 3개의 biovar로 나누어지고 있다(Cunty 등, 2015; Ferrante와 Scortichini, 2015).
본 연구에서는 우리나라에서 분리된 P. syringae pv. actinidiae 균주들의 biovar를 조사함으로써 참다래 궤양병균의 전파 경로를 이해하는 데 필요한 자료를 제공하고자 하였다.
재료 및 방법
균주 및 배양
실험에 사용된 균주는 1997년부터 1999년까지, 2008년부터 2016년 7월까지 우리나라에서 발생한 참다래 궤양병의 병징으로부터 분리되어 순천대학교 생물학과에 보관 중인 균주를 사용하였다. P. syringae pv. actinidiae F/R primers (Table 1)를 사용하여 PCR을 수행했을 때 311 bp의 DNA 절편이 증폭되는 균주를 최종적으로 P. syringae pv. actinidiae로 동정하였다(Balestra 등, 2013). 궤양병균으로 동정되어 실험에 사용된 균주 수는 총 682개였다. 궤양병 균주는 pepton-sucrose 배지(PS배지; 20 g pepton, 20 g sucrose/l)에 접종하여 28°C에서 진탕배양하였다.
DNA 추출
PS배지에서 배양한 세균 현탁액 1.5 ml를 원심분리한 뒤 제조사의 지시에 따라 Genomic DNA extraction kit (Bioneer, Daejeon, Korea)를 사용하여 세균의 유전체 DNA를 얻었다. NanoDrop One Spectrophotometer (Thermo Scientific, Waltham, MA, USA)로 DNA 농도를 측정한 뒤 –20°C에 보관하였다.
Biovar 동정
사용된 균주들의 biovar를 동정하기 위하여 PCR 방법을 사용하였다. 사용된 PCR primers의 염기서열은 Table 1과 같다. Biovar 동정에 사용된 primer들은 random amplified polymorphic DNA 분석을 통해 각 집단에 특이적인 DNA 절편으로부터 설계되었다(Koh 등, 2014; Lee 등, 2016). PsaJ-F/R primers는 biovar 1 균주에서 481 bp를, PsaK-F/R primers는 biovar 2 균주에서 413 bp를, Tac-F/R primers는 biovar 3 균주에서 545 bp의 DNA를 각각 증폭시킨다(Fig. 1). Biovar 동정용 primer를 검증하기 위하여 사용된 균주 목록은 Table 2와 같다.
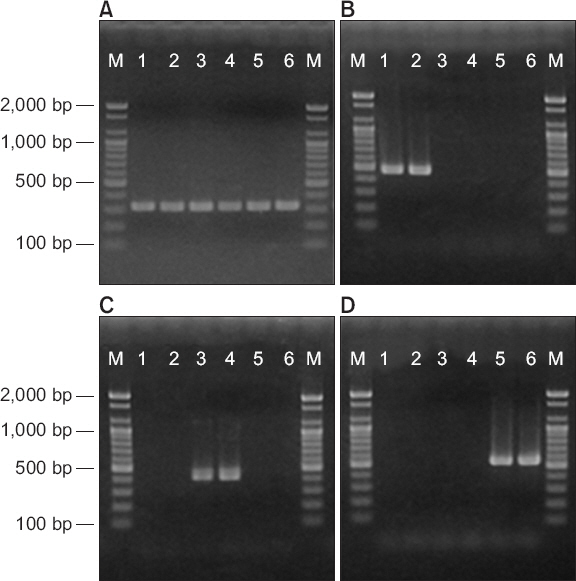
Agarose gel electrophoresis of DNA from Pseudomonas syringae pv. actinidiae strains amplified with four primer pairs; P. syringae pv. actinidiae F/R (A), PsaJ-F/R (B), PsaK-F/R (C), and Tac-F/R (D). Lane M, 1 kb DNA marker; lanes 1–2, biovar 1 strains (SUPP 1765 and NCPPB 3871); lanes 3–4, biovar 2 strains (CJW7 and KBE9); lanes 5–6, biovar 3 strains (CFBP 7286 and ICMP 18708).
PCR
PCR 반응액은 20 ng의 주형 DNA, 5 탅의 10– 반응완충액, 5 탅의 10 mM dNTP, 20 pmol의 각 primer, 2.5 U의 Top DNA polymerase (Bioneer)를 포함하여 50 탅가 되게 하였다. PCR 기기는 DNA Thermal Cycler (Takara Shuzo, Kyoto, Japan)를 사용하였으며, 반응조건은 95°C에서 5분 initial denaturation 후, 95°C에서 30초 동안 denaturation, 65°C에서 30초의 annealing, 72°C에서 30초의 extension 과정을 30회 반복하였고, 마지막으로 72°C에서 7분간의 final extension 과정을 거쳤다.
결과 및 고찰
참다래 궤양병의 병징에서 분리하여 전통적 방법을 통해 참다래 궤양병균으로 동정된 후 순천대학교 생물학과에 보관 중인 균주를 대상으로 P. syringae pv. actinidiae F/R primer를 사용한 PCR을 수행하였다. 이 primers는 궤양병균의 hopZ3 유전자 염기서열로부터 설계된 것으로, pv. actinidiae에서는 보존되어 있으나 유연관계가 가까운 pv. theae를 포함한 다른 pathovar들에서는 염기서열이 다른 부위에서 설계되었다(Balestra 등, 2013). 그 결과 총 682균주에서 예상되었던 311 bp의 DNA가 증폭되어 P. syringae pv. actinidiae인 것으로 확인되었다(Table 3).
연도별로 균주 수에서 큰 차이가 나는 것에는 특별한 의미가 없다. 본 연구진이 보관하고 있는 균주를 대상으로 조사했을 뿐이므로 연도별 균주 수와 궤양병의 발생 정도와도 관련이 없다. 2000년부터 2007년 사이에도 궤양병이 발생하였으나 그 시기에 분리된 균주가 보관되어 있지 않아 포함할 수 없었다. 다만 2014년 이후 궤양병 진단과정에서 분리된 균주는 모두 포함하였다. 균주의 수집은 전남, 경남, 제주 등 우리나라 주요 참다래 재배지에서 무작위적으로 이루어졌으므로 이 결과는 우리나라 참다래 궤양병 현황을 대표할 수 있을 것으로 본다.
전체 균주의 biovar를 결정한 결과를 Table 3에 제시하였다. 조사된 682개 균주 중 biovar 2가 288개, biovar 3가 394개 균주였다. 그러나 biovar 1에 속하는 균주는 없었다. 전체 결과 중 2013년 분리된 38개 균주에 대하여 biovar를 결정한 실험 결과를 대표로 Fig. 2에 나타내었다.
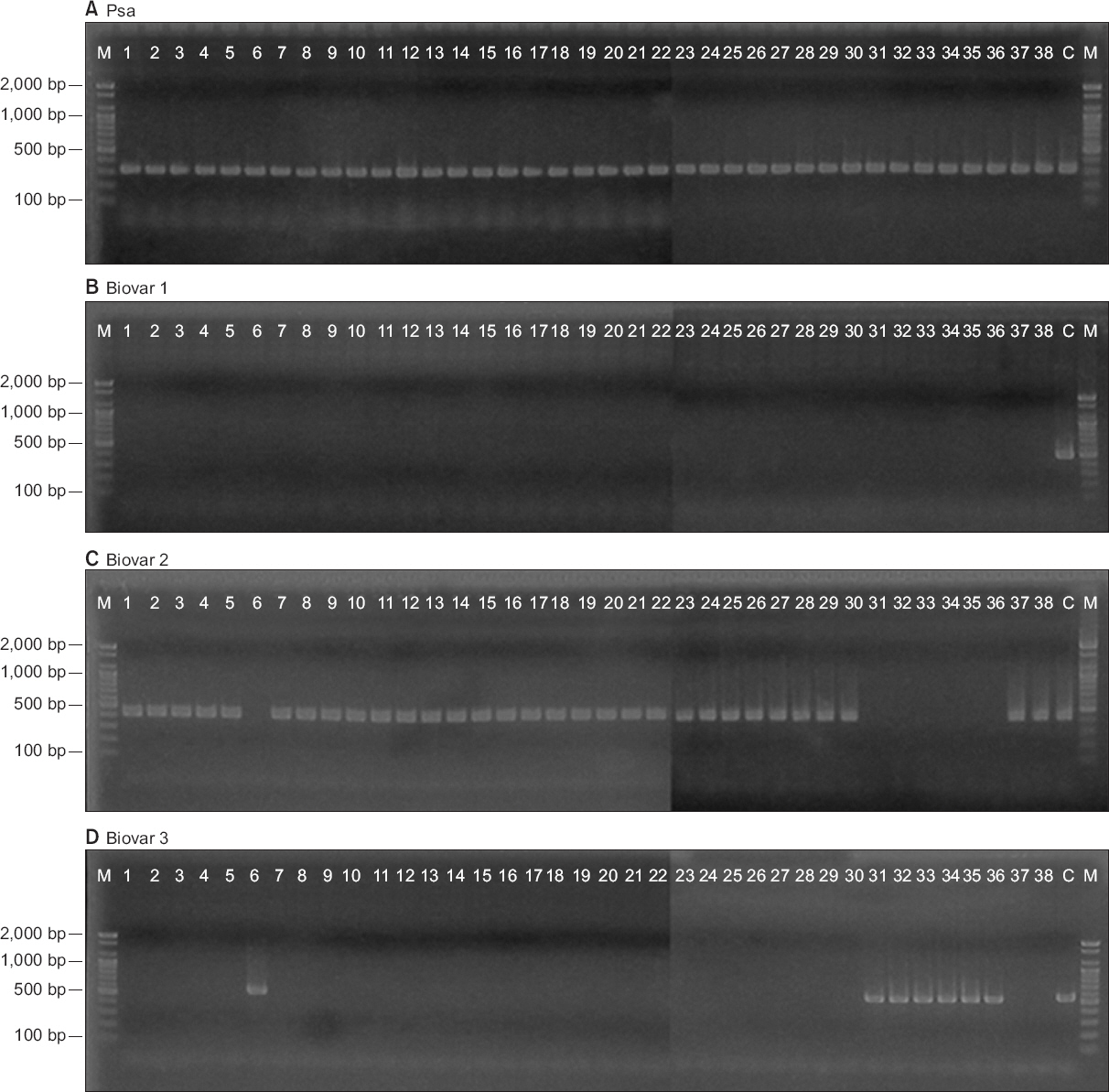
Representative results showing biovar identification of Pseudomonas syringae pv. actinidiae strains isolated in Korea. Thirty eight strains isolated in 2013 were used for amplification with four primer pairs; P. syringae pv. actinidiae F/R (A), PsaJ-F/R (B), PsaK-F/R (C), and Tac-F/R (D). Lane M, 1 kb DNA marker; lanes 1–38, P. syringae pv. actinidiae strains; lane C, positive control for each biovar.
Biovar 1은 일본에서 분리된 균주로 식물독소로 phaseolotoxin을 생산한다(Sawada 등, 1997). 우리나라에서 참다래 궤양병이 발생하기 시작했을 당시에는 궤양병균이 지리적으로 가까운 일본으로부터 유입되었을 것으로 추측하였다. 그러나 우리나라 균주는 식물독소로 phaseolotoxin을 생산하지 않고 coronatine을 생산하는 것으로 밝혀져 두 나라의 병원균이 서로 다른 집단에 속하는 것을 알게 되었고(Han 등, 2003), 유전적으로도 서로 차이가 나는 것으로 밝혀졌다(Lee 등, 2005). 지금까지 우리나라에서 biovar 1이 발견되지 않는 것으로 보아 일본균주의 유입과 전파가 일어나지 않은 것으로 보인다. 일본에서는 지금까지 알려진 biovar와 다른 새로운 MLST 그룹의 균주 집단(Psa5로 명명)이 2010년부터 발견됨으로써 biovar 1과 함께 두 종류의 집단에 의해 궤양병이 발생되고 있음이 보고된 바 있다(Sawada 등, 2014).
우리나라에서 처음 보고된 coronatine 생산 균주는 biovar 2에 속한다. Table 3에 나타난 바와 같이 biovar 2 균주는 궤양병 발생 초기부터 현재까지 지속적으로 발견되고 있다. Biovar 2에 속하는 균주가 우리나라를 제외한 다른 나라에서 발견되었다는 보고는 없다.
2008년 이탈리아에서 처음 발견된 biovar 3 균주에 대한 분자적 특성을 조사한 결과 일본 균주와는 차이가 있었으며, 특히 phaseolotoxin이나 coronatine 중 어느 것도 생산하지 않는 것이 특징이었다(Ferrante와 Scortichini, 2010). 우리나라에서는 2011년 biovar 3 균주가 처음 분리되었으며(Koh 등, 2012), 2011년 이전에 분리된 균주에서는 biovar 3가 발견되지 않았다. 현재 우리나라에서는 biovar 2와 3, 두 종류의 균주 집단이 분리되고 있다. 2011년 분리된 biovar 3에 속하는 4개 균주는 동일한 참다래 과수원에서 분리된 균주로, 2014년 9월 공적방제를 통해 폐원됨으로써 확산이 차단된 바 있다. 2014년 이후 biovar 3 균주 수의 뚜렷한 증가는 주목할 만하다. 2014년 이후 분리된 균주들의 유전자형은 2011년 균주와 다른 것으로 보아 외부로부터의 유입이 있었을 것으로 추측할 수 있다. 실제로 2014년 이후 biovar 3에 의한 궤양병의 급격한 증가는 외국에서 수입된 오염된 꽃가루에 의한 것으로 판명되고 있다(Kim 등, 2016).
우리나라에서 biovar 3에 의한 궤양병의 빠른 확산이 이 균주의 특성에 기인한다고 추측할 수 있다. 지금까지 각 biovar에 따른 병원성을 비교한 연구는 없다. 다만 이탈리아의 예를 통해 간접적으로 비교할 수 있다. Biovar 1 균주는 일본에서 심각한 피해를 주었음에도 이탈리아에서는 1992년 이래 약 20년간 간헐적으로 발생하였을 뿐이었고 피해도 크지 않았다(Ferrante와 Scortichini, 2010). 그러나 biovar 3가 2008년 Latium 지방에서 처음 출현한 이래 2–3년 사이 이탈리아 전체 참다래 재배 지역에서 이 균주에 의한 궤양병이 발생하였고, 감염된 과수원을 폐원시킬 만큼 심각한 피해를 일으켰다(Marcelletti와 Scortichini, 2011). 이러한 사실은 biovar 3가 빠른 전파력을 가지고 있을 뿐 아니라 병원성도 강하다는 것을 의미한다.
2008년 이후 biovar 3가 문제되고 있는 나라들에서 biovar 1이나 biovar 2가 함께 발견되었다는 보고는 없다. 그 이유 중 하나는 우리나라와 일본의 참다래 생산량은 자체 소비량을 충당하지 못할 정도여서 많은 부분을 수입에 의존하고 있다. 따라서 두 나라로부터 다른 나라로 과일, 꽃가루 및 묘목 등 식물체의 공식적인 유출은 없기 때문인 것으로 생각된다.
Biovar 3 집단에 속한 균주들 사이에서도 지리적 기원에 따른 유전적 다양성이 확인됨에 따라(Ciarroni 등, 2015) 각 아집단(subpopulation)의 분자마커가 개발되고 있다(Balestra 등, 2013). 병원균 집단의 구조가 밝혀지고 각 집단을 검출할 수 있는 분자마커가 개발되면 이를 통해 전 세계적 차원에서 biovar 3의 이동과 확산 경로를 파악할 수 있을 것이다.
요약
Pseudomonas syringae pv. actinidiae는 참다래에 궤양병을 일으키는 원인세균이다. 이 병원균 집단은 분자적 특성에 따라 biovar 1, 2, 3로 나누어진다. 본 연구에서는 1997년부터 우리나라에서 분리되어 순천대학교 생물학과에 보관 중인 P. syringae pv. actinidiae 균주들의 biovar를 기존에 발표된 biovar에 특이적인 PCR primers를 사용하여 동정하였다. 전체 682개 균주 중 biovar 2에 속한 균주가 288개, biovar 3에 속한 균주가 394개였다. 그러나 우리나라에서 분리된 균주 중 biovar 1에 속하는 균주는 없었다. Biovar 3 균주에 의한 궤양병의 갑작스러운 발생과 확산은 이 균주가 빠른 전파력을 가지고 있음을 말해 준다.
Acknowledgement
This work was supported by the Korea Institute of Planning and Evaluation for Technology in Food, Agriculture, Forestry and Fisheries (IPET) through Agri-Bio Industry Technology Development Program, funded by Ministry of Agriculture, Food, and Rural Affairs (MAFRA) (315019-02-1-SB020).
References
Notes
Conflicts of Interest
No potential conflict of interest relevant to this article was reported.