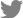|
 |
| Res. Plant Dis > Volume 26(4); 2020 > Article |
|
ABSTRACT
We have developed a simultaneous diagnostic method that can identify both the species of thrips and tomato spotted wilt virus (TSWV) that are problematic in chrysanthemum plants. This is a method of amplifying DNA by performing reverse transcription-polymerase chain reaction by simultaneously adding primers specific to TSWV coat protein (N) gene and primers specific to the internal transcribed spacer 2 region of Frankliniella occidentalis and F. intonsa using total nucleic acid extracted from one thrips. The sizes of DNA fragments for TSWV, F. occidentalis, and F. intonsa were 777, 287, and 367 bp, respectively. These results showed species identification of thrips and whether thrips carrying TSWV can be simultaneously confirmed. Further usefulness of the simultaneous diagnostic method was made from greenhouse survey at chrysanthemum greenhouses in Taean (Chungcheongnam-do) and Changwon (Gyeongsangnam-do) to investigate the identification of thrips species and the rate of thrips carrying TSWV. Of thrips collected from the greenhouses, 83.7% thrips was F. occidentalis and 72.9% F. occidentalis carried TSWV in Taean. Similarly, the diagnostic method showed that 92.2% thrips was F. occidentalis and 84.0% F. occidentalis carried TSWV in Changwon. These results confirm that F. occidentalis is a dominant thrips species and the thrips species plays a crucial role in the transmission of TSWV in chrysanthemum plants in the greenhouses. Taken together, this study showed a simple diagnostic method for thrips identification and epidemiological studies of the timing and spread of TSWV through thrips in chrysanthemum greenhouses in South Korea.
ĻĄŁĒÖöļŖö ĻĄŁĒÖöĻ│╝ņŚÉ ņåŹĒĢśļŖö ņŚ¼ļ¤¼ĒĢ┤ņé┤ņØ┤ ņŗØļ¼╝ļĪ£ Ļ┤ĆņāüņÜ®, ņĢĮņÜ®Ļ│╝ ņŗØņÜ®ņ£╝ļĪ£ ĒÖ£ņÜ®ļÉ£ļŗż. ĻĄŁĒÖöļŖö ņŖżĒāĀļŗżļō£ ĻĄŁĒÖö(ļīĆĻĄŁ)Ļ│╝ ņŖżĒöäļĀłņØ┤ ĻĄŁĒÖö(ņåīĻĄŁ)ņ£╝ļĪ£ ĻĄ¼ļČäĒĢĀ ņłś ņ׳ņ£╝ļ®░, ĻĄŁļé┤ņŚÉņä£ļŖö ņŖżĒāĀļŗżļō£ ĻĄŁĒÖöņØś ņ×¼ļ░░ļ®┤ņĀü(242.9 ha)ņØ┤ ņĀäņ▓┤ ĻĄŁĒÖö ņ×¼ļ░░ļ®┤ņĀü(309.1 ha)ņØś 78.6% ļź╝ ņ░©ņ¦ĆĒĢ£ļŗż(Ministry of Agriculture, Food and Rural Affairs, 2020). ĻĄŁļé┤ ņ×¼ļ░░ņ¦Ć Ļ░ĆņÜ┤ļŹ░ Ļ▓Įļé© ņ░ĮņøÉ ļ░Å ļČĆņé░ ņ¦ĆņŚŁņØĆ ņŗĀļ¦ł, ļ░▒ļ¦ł, ļ░▒ņäĀ ĒÆłņóģ ļō▒ņØä ņŖżĒāĀļŗżļō£ ĻĄŁĒÖöļź╝ ņŗ£ņäżņŚÉņä£ ņØ╝ļ│Ė ņłśņČ£ņÜ®ņ£╝ļĪ£ ņ×¼ļ░░ĒĢśĻ│Ā ņ׳ņ£╝ļ®░ ĻĘĖ ņ×¼ļ░░ļ®┤ņĀüņØĆ 125.5 haņŚÉ ļŗ¼ĒĢ£ļŗż(Ministry of Agriculture, Food and Rural Affairs, 2020).
ĻĄŁĒÖö ņ×¼ļ░░ņŚÉņä£ ņŻ╝ļĪ£ ļ¼ĖņĀ£Ļ░Ć ļÉśļŖö Orthotospovirus ņåŹņŚÉ ņåŹĒĢśļŖö ļ░öņØ┤ļ¤¼ņŖżļōżņØĆ ĒåĀļ¦łĒåĀļ░śņĀÉņ£äņĪ░ļ░öņØ┤ļ¤¼ņŖż(Tomato spotted wilt virus, TSWV)ņÖĆ ĻĄŁĒÖöņżäĻĖ░Ļ┤┤ņĀĆļ░öņØ┤ļ¤¼ņŖż(Chrysanthemum stem necrosis virus, CSNV)ļĪ£ ņĢīļĀżņĀĖ ņ׳ļŗż(Chung ļō▒, 2006; Yoon ļō▒, 2017). TSWVņÖĆ CSNVļŖö 3Ļ░£ņØś (ŌĆÆ) stranded Ēś╣ņØĆ ambisense RNAņØś ļČäņĀł Ļ▓īļåł(L, M ļ░Å S segments) ĒśĢĒā£ļĪ£ ņØ┤ļŻ©ņ¢┤ņĀĖ ņ׳ņ£╝ļ®░, ņŗØļ¼╝Ļ│╝ ņŗØļ¼╝ ņé¼ņØ┤ņØś ņĀäņŚ╝ņØĆ ņŻ╝ļĪ£ ņ┤Øņ▒äļ▓īļĀłņŚÉ ņØśĒĢ┤ ļ¦żĻ░£ļÉśņ¢┤ ĻĄŁĒÖö, Ļ│ĀņČö, ĒåĀļ¦łĒåĀ, ņ×Īņ┤łļźś ļō▒ 1,090ņŚ¼ņóģ ņŗØļ¼╝ņØä Ļ░ÉņŚ╝ņŗ£Ēé©ļŗż(Parrella ļō▒, 2003). ĻĄŁļé┤ņŚÉņä£ TSWVļŖö 2003ļģä ņśłņé░ņ¦ĆņŚŁ ĒīīĒöäļ”¼ņ╣┤ ņ×¼ļ░░ļåŹĻ░ĆņŚÉņä£ ņ▓śņØī ļ░£Ļ▓¼ļÉ£ ņØ┤Ēøä, ļŗżņ¢æĒĢ£ ņøÉņśłņ×æļ¼╝ņŚÉņä£ Ēö╝ĒĢ┤Ļ░Ć ĒÖĢņØĖļÉśņŚłņ£╝ļ®░ ĻĄŁĒÖöņŚÉņä£ļŖö 2006ļģä ņČ®ļé© Ēā£ņĢł ļ░Å ņśłņé░ņØś ĻĄŁĒÖö ņŗ£ņäżĒĢśņÜ░ņŖż(ĒÆłņóģ: ņŗĀļ¦ł, ĒÖöļ×æ)ņŚÉņä£ TSWVĻ░Ć ņ▓śņØī ļ│┤Ļ│ĀļÉśņŚłļŗż(Chung ļō▒, 2006; Kim ļō▒, 2004).
ņĀä ņäĖĻ│äņĀüņ£╝ļĪ£ ĻĄŁĒÖö ņ×¼ļ░░ņŚÉņä£ ļ¼ĖņĀ£ļÉśļŖö ĒĢ┤ņČ®ņØĆ ņ┤Øņ▒äļ▓īļĀł, ņ¦äļö¦ļ¼╝, ņØæņĢĀ ļō▒ņØ┤ļ®░ ĻĘĖ ņżæ Ļ░Ćņן Ēö╝ĒĢ┤ļź╝ ņŻ╝ļŖö ĒĢ┤ņČ®ņØĆ ņ┤Øņ▒äļ▓īļĀłļĪ£ ļ│┤Ļ│ĀļÉśņŚłļŗż(Lewis, 1997). ņ┤Øņ▒äļ▓īļĀłļŖö ņäĖĻ│äņĀüņ£╝ļĪ£ ņĢĮ 5,500ņŚ¼ņóģņØ┤ ļ│┤Ļ│ĀļÉśņŚłņ£╝ļ®░ 87ņóģņØ┤ Ļ▓ĮņĀ£ņĀüņ£╝ļĪ£ ņżæņÜöĒĢ£ ņ×æļ¼╝ņŚÉ Ēö╝ĒĢ┤ļź╝ ņŻ╝Ļ│Ā ņ׳ļŖö Ļ▓āņ£╝ļĪ£ ļ│┤Ļ│ĀļÉśņŚłļŗż(Demirozer ļō▒, 2012). ĻĄŁļé┤ņŚÉņä£ļŖö ĻĄŁĒÖö, Ļ│ĀņČö, Ēīī, ņśżņØ┤ ļō▒ņŚÉņä£ Ļ▓ĮņĀ£ņĀüņ£╝ļĪ£ Ēü░ Ēö╝ĒĢ┤ļź╝ ņŻ╝ļŖö ņ┤Øņ▒äļ▓īļĀł ņóģļźśļĪ£ļŖö Ēīīņ┤Øņ▒äļ▓īļĀł(Thrips tabaci), ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł(Frankliniella occidentalis), ļ│╝ļĪØņ┤Øņ▒äļ▓īļĀł(Scirtothrips dorsalis) ļ░Å ņśżņØ┤ņ┤Øņ▒äļ▓īļĀł(T. palmi)ļĪ£ ļ│┤Ļ│ĀļÉśņŚłņ£╝ļ®░, ņĢ×ņä£ ĻĖ░ņłĀļÉ£ ņ┤Øņ▒äļ▓īļĀł ņóģņØä ĒżĒĢ©ĒĢśņŚ¼ TSWVļź╝ ļ¦żĻ░£ĒĢśļŖö ņ┤Øņ▒äļ▓īļĀłļŖö 11ņóģņØ┤ ļ│┤Ļ│ĀļÉśņŚłļŗż(Hyun ļō▒, 2012; MorseņÖĆ Hoddle, 2006). ņØ┤ļōż ņ┤Øņ▒äļ▓īļĀłļźśļŖö ņé┤ņČ®ņĀ£ņŚÉ Ļ░ĢĒĢ£ ņĢĮņĀ£ ņĀĆĒĢŁņä▒ņØä Ļ░Ćņ¦Ćļ®░, ņ×Ä ļÆĘļ®┤ ļśÉļŖö ĻĮā ņåŹņŚÉ ņł©ļŖö ĒŖ╣ņä▒ ļĢīļ¼ĖņŚÉ ņĢĮņĀ£ ļ░®ņĀ£Ļ░Ć ņ¢┤ļĀżņÜ┤ ĒĢ┤ņČ®ņ£╝ļĪ£ ņĢīļĀżņĀĖ ņ׳ļŗż(Han ļō▒, 1998; Lee, 1996; SchmidtņÖĆ Frey, 1995). ņä▒ņČ®ņØĆ ĒÖ®ņāē ļśÉļŖö ĒÖ®Ļ░łņāēņØä ļØäļ®░ ĒÅēĻĘĀ Ēü¼ĻĖ░ļŖö 1.17 mm ļé┤ņÖĖņØ┤ļ®░ ņŗØļ¼╝ņ▓┤ ņĪ░ņ¦ü ņåŹņŚÉ ņé░ļ×ĆĒĢśļŖö Ļ▓āņ£╝ļĪ£ ņĢīļĀżņĀĖ ņ׳ļŗż. ņ×ÄņŚÉņä£ ļČĆĒÖöĒĢ£ ņĢīņŚÉņä£ 1ļĀ╣ ņĢĮņČ® ļŗ©Ļ│äļź╝ Ļ▒░ņ│É 2ļĀ╣ ņĢĮņČ® ņäĖļīĆļź╝ Ļ▒░ņ╣śļ®░ ņØ┤Ēøä ņŻ╝ļĪ£ ĒåĀņ¢æņ£╝ļĪ£ ņØ┤ļÅÖĒĢśņŚ¼ ĒåĀņ¢æņŚÉņä£ ļ▓łļŹ░ĻĖ░ Ļ│╝ņĀĢ(ņĀäņØśņÜ®, ĒøäņØśņÜ®)ņØä Ļ▒░ņ╣£ ļŗżņØī ņä▒ņČ®ņ£╝ļĪ£ ņÜ░ĒÖö Ēøä ņ¦ĆņāüļČĆļĪ£ ņØ┤ļÅÖĒĢśņŚ¼ ņŗØļ¼╝ņØä Ļ░ĆĒĢ┤ĒĢ£ļŗż. ĻĄŁĒÖöņŚÉņä£ļŖö ļ¼ĖņĀ£ļÉśļŖö ņ┤Øņ▒äļ▓īļĀłļźśļŖö ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłņÖĆ ļīĆļ¦īņ┤Øņ▒äļ▓īļĀł(F. intonsa)ņ£╝ļĪ£ ņĢīļĀżņĀĖņ׳ņ£╝ļéś(OkazakiņÖĆ Sakurai, 2005; Okuda ļō▒ 2013), ļģĖņ¦Ćņ×¼ļ░░ņŚÉ ļ╣äĒĢ┤ ņŗ£ņäżņ×¼ļ░░ ĻĄŁĒÖöņŚÉņä£ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłĻ░Ć ļåÆņØĆ ļ░ĆļÅäļĪ£ ļ░£ņāØļÉśĻ│Ā ņ׳ļŗż(Park ļō▒, 2002; Yoon ļō▒, 2020). ņ┤Øņ▒äļ▓īļĀłļŖö ņŻ╝ļĪ£ ņ¢┤ļ”░ ĻĄŁĒÖö ņ×ÄņØä Ļ░ĆĒĢ┤ĒĢśņŚ¼ ņ×ÄņØś ĻĖ░ĒśĢņØä ņ┤łļלĒĢśĻ│Ā, ĻĄŁĒÖö ĻĮāņØ┤ Ēö╝ĻĖ░ ņŗ£ņ×æĒĢśļ®┤ ņ£ĀņČ®Ļ│╝ ņä▒ņČ®ļōżņØ┤ ĻĮā ņĢłņ£╝ļĪ£ ļōżņ¢┤Ļ░Ć ņ¢┤ļ”░ ĻĮāņ×ÄņØä Ļ░ēņĢä ņāüņ▓śļź╝ ļéĖļŗż. ņØ┤Ēøä ĻĮāņ×ÄņØ┤ ņĀäĻ░£ļÉśļ®┤ ņāüņ▓śļČĆņ£äĻ░Ć ņŗ£ļōżĻ│Ā ĒāłņāēļÉśņ¢┤ ĻĄŁĒÖöņØś ņāüĒÆł Ļ░Ćņ╣śļź╝ ļ¢©ņ¢┤ļ£©ļ”¼Ļ▓ī ļÉ£ļŗż. ņØ┤ļ¤¼ĒĢ£ ņ┤Øņ▒äļ▓īļĀłļŖö TSWV ļ░Å CSNVļź╝ ļ¦żĻ░£ĒĢ£ļŗż. ņ┤Øņ▒äļ▓īļĀłļŖö 1ŌĆÆ2ļĀ╣ ņĢĮņČ® ņŗ£ĻĖ░ņŚÉ TSWVļź╝ ĒØĪņ”ÖĒĢśņŚ¼ ņśüņåŹņĀäņŚ╝(persistent transmission)ņ£╝ļĪ£ ņĀäņŚ╝, ĒÖĢņé░ņŗ£ĒéżļŖö Ļ▓āņ£╝ļĪ£ ņĢīļĀżņĀĖ ņ׳ņ£╝ļéś ļŗżļźĖ ņ┤Øņ▒äļ▓īļĀł ņóģĻ│╝ ļŗżļź┤Ļ▓ī ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłņÖĆ ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłļŖö ņä▒ņČ® ļŗ©Ļ│äņŚÉņä£ļÅä TSWVļź╝ ņĀäņŚ╝ņŗ£Ēé©ļŗż(Inoue ļō▒ 2004; Maris, 2004; Wijkamp ļō▒, 1996).
ņ┤Øņ▒äļ▓īļĀł ņóģ ļÅÖņĀĢļ▓Ģņ£╝ļĪ£ internal transcribed spacer 2 (ITS2) ņśüņŚŁņØś PCR ņé░ļ¼╝ņØä ņĀ£ĒĢ£ĒÜ©ņåī RsaIņ£╝ļĪ£ ņ▓śļ”¼ĒĢśļŖö polymerase chain reactionŌĆÆrestriction fragment length polymorphism ļ▓Ģ ļ░Å ITS2 ņ£ĀņĀäņ×ÉņØś ņóģ ĒŖ╣ņØ┤ņĀüņØĖ ĒöäļØ╝ņØ┤ļ©Ėļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ PCR ļ░Å ņŚ╝ĻĖ░ņä£ņŚ┤ ļČäņäØņØä ņłśĒ¢ēĒĢśļŖö ļ░®ļ▓Ģ ļō▒ņØ┤ Ļ░£ļ░£ļÉśņ¢┤ ņ׳ļŗż(Brunner ļō▒, 2002; NakaharaņÖĆ Minoura, 2015; TodaņÖĆ Komazaki, 2002; Yeh ļō▒, 2014, 2015). ņØ╝ļ░śņĀüņ£╝ļĪ£ Ļ░ÉņŚ╝ļÉ£ ņŗØļ¼╝ņ▓┤ņŚÉņä£ TSWVļź╝ Ļ▓ĆņČ£ĒĢśĻĖ░ ņ£äĒĢ┤ ĒÜ©ņåīĻ▓░ĒĢ®ļ®┤ņŚŁĒØĪņ░®Ļ▓Ćņé¼ļ▓Ģ(enzyme-linked immunosorbent assay)ņÖĆ ņŚŁņĀäņé¼ņżæĒĢ®ĒÜ©ņåīņŚ░ņćäļ░śņØæļ▓Ģ(reverse transcriptionŌĆÆpolymerase chain reaction, RT-PCR), ņĀĢļ¤ēņĀü ņŚŁņĀäņé¼ņżæĒĢ®ĒÜ©ņåīņŚ░ņćäļ░śņØæļ▓Ģ(quantitative transcriptionŌĆÆpolymerase chain reaction)ņØ┤ ļ│┤Ļ│ĀļÉśņŚłļŗż(Chung ļō▒, 2006, Yoon ļō▒, 2017). ļśÉĒĢ£ ņ┤Øņ▒äļ▓īļĀłņŚÉņä£ TSWVļź╝ Ļ▓ĆņČ£ĒĢśĻĖ░ ņ£äĒĢśņŚ¼ ņĢ×ņä£ ĻĖ░ņłĀĒĢ£ 3Ļ░Ćņ¦Ć ļ░®ļ▓ĢļōżņØä ņØ┤ņÜ® ĻĖ░ņłĀņØ┤ ļ│┤Ļ│ĀļÉśņŚłņ£╝ļéś ņØ┤ļ¤¼ĒĢ£ ļ░®ļ▓ĢņØĆ ņŗ£Ļ░äņØ┤ ļ¦ÄņØ┤ ņåīņÜöļÉśĻ▒░ļéś ļ»╝Ļ░ÉļÅäĻ░Ć ļ¢©ņ¢┤ņ¦ĆĻ│Ā ĻĄ¼ņ×ģ ņןļ╣äĻ░Ć ļ╣äņŗĖļŗżļŖö ļŗ©ņĀÉņØ┤ ņ׳ņ£╝ļ®░ ļśÉĒĢ£ ņ┤Øņ▒äļ▓īļĀł ņóģ ļÅÖņĀĢņØä ļ│äļÅäļĪ£ ņłśĒ¢ēĒĢ┤ņĢ╝ ĒĢśļŖö ļ▓łĻ▒░ļĪ£ņøĆņØ┤ ņ׳ļŗż(Bandla ļō▒, 1994; Boonham ļō▒, 2002; Cho ļō▒, 1989; Tsuda ļō▒, 1994).
ņØ┤ļ▓ł ņŚ░ĻĄ¼ņŚÉņä£ TSWV ņÖĖĒö╝ļŗ©ļ░▒ņ¦ł(N) ņ£ĀņĀäņ×É ļ░Å ņ┤Øņ▒äļ▓īļĀł ITS2 ļČĆļČäņŚÉ ĒŖ╣ņØ┤ņĀüņØĖ ĒöäļØ╝ņØ┤ļ©Ėļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ņ┤Øņ▒äļ▓īļĀł 1ļ¦łļ”¼ņŚÉņä£ TSWV ļ│┤ļÅģ ņŚ¼ļČĆ ļ░Å ņ┤Øņ▒äļ▓īļĀłņØś ņóģ ļÅÖņĀĢņØä ļÅÖņŗ£ņŚÉ ĒÖĢņØĖĒĢĀ ņłś ņ׳ļŖö ņ¦äļŗ©ļ▓ĢņØä Ļ░£ļ░£ĒĢśņśĆļŗż. ņØ┤ļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ĻĄŁĒÖö ņ×¼ļ░░ ņś©ņŗżņŚÉņä£ ņłśņ¦æĒĢ£ ņ┤Øņ▒äļ▓īļĀłņŚÉņä£ TSWV ļ│┤ļÅģļźĀ ļ░Å ņ┤Øņ▒äļ▓īļĀł ņÜ░ņĀÉņóģņØä ļÅÖņŗ£ņŚÉ ņĪ░ņé¼Ļ░Ć Ļ░ĆļŖźĒĢ©ņØä ļ│┤ņŚ¼ņŻ╝ļ®░ ņ┤Øņ▒äļ▓īļĀłļź╝ ĒåĄĒĢ£ TSWVņØś ņŗ£ņäżļé┤ ņ£Āņ×ģņŗ£ĻĖ░ņÖĆ ļ░£ņāØ Ļ▓ĮļĪ£ ļō▒ TSWV ņŚŁĒĢÖ ņŚ░ĻĄ¼ņŚÉ ĒÖ£ņÜ®ĒĢĀ ņłś ņ׳ņØīņØä ņśłņŗ£ĒĢ┤ņżĆļŗż. ņØ┤ļ¤¼ĒĢ£ ņŚ░ĻĄ¼ļŖö ņŗ£ņäżĻĄŁĒÖöņŚÉņä£ TSWV ļ│æņ¦ĢņØ┤ ļéśĒāĆļéśĻĖ░ ņØ┤ņĀä ņ┤Øņ▒äļ▓īļĀłļź╝ ņØ┤ņÜ®ĒĢ£ TSWV ņśłņ░░ņŚÉ ĒÖ£ņÜ®ĒĢĀ ņłś ņ׳ņØä Ļ▓āņØ┤ļØ╝ ņāØĻ░üļÉ£ļŗż.
Ļ▓Įļé© ņ░ĮņøÉņŗ£ ĻĄŁĒÖöņ×¼ļ░░ ņŗ£ņäż ĒĢśņÜ░ņŖżņŚÉņä£ ņłśņ¦æĒĢ£ TSWV Ļ░ÉņŚ╝ ĻĄŁĒÖö(ļ░▒ļ¦ł ĒÆłņóģ)ļź╝ Ļ│Ąņŗ£ņ×¼ļŻīļĪ£ ņé¼ņÜ®ĒĢśņśĆļŗż. TSWV Ļ░ÉņŚ╝ ĻĄŁĒÖöļŖö ņāüņŚĮĻ│╝ ņżäĻĖ░Ļ░Ć ļÆżĒŗĆņ¢┤ņĀĖ ņ׳Ļ│Ā ļ│æņØ┤ ņ¦äņĀäļÉśļ®┤ ņ×ÄĻ│╝ ņżäĻĖ░ ļüØņŚÉ Ļ┤┤ņé¼ņ”ØņāüņØ┤ ļéśĒāĆļéś Ļ│Āņé¼ĒĢśņśĆļŗż(Fig. 1). TSWV Ļ░ÉņŚ╝ ņĪ░ņé¼ļŖö SEB1 ņÖäņČ®ņĢĪ(Agdia, Beltsville, MD, USA)ņŚÉ ņ£äņØś ĻĄŁĒÖöņØś ļ│æļōĀ ņ×ÄņØä ļ¦łņćäĒĢ£ ņĪ░ņ”ÖņĢĪņŚÉ TSWV ĒŖ╣ņØ┤ņĀü ImmunoStrip (Agdia)ņØä ļŗ┤ĻĘ╝ Ēøä ļéśĒāĆļéśļŖö Ļ▓░Ļ│╝ļĪ£ ĒÖĢņØĖĒĢśņśĆļŗż. TSWVĻ░Ć ņ¦äļŗ©ļÉ£ ĻĄŁĒÖö ņ×ÄņØä 0.1 M potassium phosphate (pH 7.2) ņÖäņČ®ņĢĪņŚÉņä£ ļ¦łņćäĒĢ£ ņĪ░ņ”ÖņĢĪņØä carborundum (Thermo Fisher Scientific, Waltham, MA, USA)ņØä ļ┐īļ”░ ņ▓Łņ¢æĻ│ĀņČö(Capsicum annuum L.)ņŚÉ ņĀæņóģĒĢśņśĆļŗż. 2ņŻ╝ Ēøä ņāüņŚĮņŚÉ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ļ░Å ļīĆļ¦īņ┤Øņ▒äļ▓īļĀł 1ŌĆÆ2ļĀ╣ ņĢĮņČ®ņØä ņŗØļ¼╝ņ▓┤ļŗ╣ 30ļ¦łļ”¼ņö® ļČōņØä ņØ┤ņÜ®ĒĢśņŚ¼ ņś¼ļĀż TSWVļź╝ ļ│┤ļÅģņŗ£Ēé© ļŗżņØī 7ņØ╝ ņØ┤ņāü ņ£Āņ¦Ćņŗ£ņ╝£ ņä▒ņČ®ņ£╝ļĪ£ ņ£ĀļÅäļÉ£ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ļśÉļŖö ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłļź╝ ņØ┤Ēøä ņŗżĒŚśņŚÉ ņé¼ņÜ®ĒĢśņśĆļŗż.
Ļ░£ļ░£ĒĢ£ ĻĖ░ņłĀņØä ņ”Øļ¬ģĒĢśĻĖ░ ņ£äĒĢśņŚ¼ Ļ▓Įļé© ņ░ĮņøÉņŗ£ ļ░Å ņČ®ļé© Ēā£ņĢłņ¦ĆņŚŁ ĻĄŁĒÖö ņ×¼ļ░░ņ¦ĆņŚÉņä£ ņ┤Øņ▒äļ▓īļĀłļź╝ Ļ░ü 43, 102ļ¦łļ”¼ļź╝ Ēżņ¦æĒĢśņśĆņ£╝ļ®░ Ļ░ü ņ┤Øņ▒äļ▓īļĀłļź╝ ņŗżņ▓┤Ēśäļ»ĖĻ▓Į(Carl Zeiss, Jena, Germany)ņØä ņØ┤ņÜ®ĒĢśņŚ¼ Ļ░äņØ┤ ļÅÖņĀĢņØä ņłśĒ¢ēĒĢśņśĆĻ│Ā Ļ░£ņ▓┤ļ│äļĪ£ 70% ņŚÉĒāäņś¼ņØ┤ ļŗ┤ĻĖ┤ 1.5 ml Eppendorf tubeņŚÉ ļäŻņ¢┤ ŌłÆ20┬░CņŚÉ ļ│┤Ļ┤ĆĒĢśĻ▒░ļéś ĒĢĄņé░ ņČöņČ£ņŚÉ ņé¼ņÜ®ĒĢśņśĆļŗż.
ĒĢĄņé░ņČöņČ£ņØä ņ£äĒĢ┤ ņ”ØņāüņØ┤ ļ│┤ņØ┤ļŖö ĻĄŁĒÖö ņ×Ä 100 mg Ēś╣ņØĆ ņ┤Øņ▒äļ▓īļĀł 1ŌĆÆ10ļ¦łļ”¼(0.05ŌĆÆ0.5 mg)ļź╝ ņĢĪņ▓┤ņ¦łņåīĻ░Ć ļōżņ¢┤ ņ׳ļŖö 1.5 ml Eppendorf tubeņŚÉ ļäŻņØĆ ļŗżņØī ļ¦łņćäĒĢśņśĆļŗż. ņØ┤Ēøä ņĀ£ņĪ░ņé¼ņØś ņČöņ▓£ ļ░®ļ▓ĢļīĆļĪ£ Plant RNA/DNA purification kit (Norgenbiotek, Thorold, ON, Canada)ļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ĒĢĄņé░ņØä ņČöņČ£ĒĢśņśĆļŗż. ņČöņČ£ļÉ£ ĒĢĄņé░ņØś ņł£ļÅäļŖö NanoDrop ļČäĻ┤æĻ┤æļÅäĻ│ä(Thermo Fisher Scientific)ļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ĒØĪĻ┤æļÅä 260 nmņÖĆ 280 nm ļ╣äņ£© (A260/A280)ļĪ£ Ļ▓░ņĀĢĒĢśņśĆļŗż. ņČöņČ£ļÉ£ ĒĢĄņé░ņØĆ ņŗżĒŚśņŚÉ ļ░öļĪ£ ņé¼ņÜ®ĒĢśĻ▒░ļéś ŌĆÆ80┬░C ļāēļÅÖĻ│ĀņŚÉ ļ│┤Ļ┤ĆĒĢśņŚ¼ ņØ┤Ēøä ņŗżĒŚśņŚÉ ņé¼ņÜ®ĒĢśņśĆļŗż.
ņ┤Øņ▒äļ▓īļĀłņØś ņóģ ļÅÖņĀĢ ļ░Å ļ│┤ļÅģĒĢśĻ│Ā ņ׳ļŖö TSWVņØś ļŗ©ļÅģ Ēś╣ņØĆ ļÅÖņŗ£ņ¦äļŗ©ņØś Ļ░£ļץņĀü ļ░®ļ▓ĢņØĆ Fig. 2AņŚÉ ņĀ£ņŗ£ļÉśņŚłļŗż. ņ┤Øņ▒äļ▓īļĀłņØś ņóģ ļÅÖņĀĢ ļ░Å ļ│┤ļÅģĒĢśĻ│Ā ņ׳ļŖö TSWVņØś ļŗ©ļÅģ Ēś╣ņØĆ ļÅÖņŗ£ņ¦äļŗ©ņØĆ ĒĢĄņé░ ņČöņČ£ņØä ņØ┤ņÜ®ĒĢśņŚ¼ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ļ░Å ļīĆļ¦īņ┤Øņ▒äļ▓īļĀł ITS2 ļČĆņ£äņŚÉ ĒŖ╣ņØ┤ņĀüņØĖ ĒöäļØ╝ņØ┤ļ©Ė(3ŌĆ▓ ļ¦Éļŗ©ļČĆņŚÉ ņŚŁļ░®Ē¢ź ĒöäļØ╝ņØ┤ļ©ĖļĪ£ņä£ Ļ│ĄĒåĄņĀüņ£╝ļĪ£ ThripsITS2R3ņØ┤ ņé¼ņÜ®ļÉśņŚłņ£╝ļ®░ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłļŖö OCC-ITS2F6, ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłļŖö INT-ITSF1ņØä ņĀĢļ░®Ē¢ź ĒöäļØ╝ņØ┤ļ©ĖļĪ£ ņé¼ņÜ®)ļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ RT-PCRļĪ£ ņłśĒ¢ēĒĢśņśĆļŗż(Nakahara ņÖĆ Minoura, 2015). ĻĄŁĒÖö ļ░Å ņ┤Øņ▒äļ▓īļĀłņŚÉņä£ TSWVļź╝ ņ¦äļŗ©ĒĢśĻĖ░ ņ£äĒĢśņŚ¼ TSWVņØś S RNAņŚÉ ņĢöĒśĖĒÖöļÉśņ¢┤ ņ׳ļŖö ņÖĖĒö╝ļŗ©ļ░▒ņ¦ł ņ£ĀņĀäņ×É(N ņ£ĀņĀäņ×É)ļź╝ ĒŖ╣ņØ┤ņĀüņ£╝ļĪ£ ņ”ØĒÅŁĒĢśļÅäļĪØ Ļ│ĀņĢłļÉ£ ĒöäļØ╝ņØ┤ļ©Ė(TSWV-NCP-For ļ░Å TSWV-NCP-Rev)ļź╝ ņØ┤ņÜ®ĒĢśņśĆļŗż(Table 1) (Yoon ļō▒, 2014).
ņČöņČ£ļÉ£ ĒĢĄņé░ 10 ngĻ│╝ TSWV ņ¦äļŗ© ĒöäļØ╝ņØ┤ļ©Ė ņäĖĒŖĖ(TSWV-NCP-For ļ░Å TSWV-NCP-Rev) ļ░Å ņ┤Øņ▒äļ▓īļĀł ņ¦äļŗ© ĒöäļØ╝ņØ┤ļ©Ė ņäĖĒŖĖ(OCC-ITS2F6ņÖĆ ThripsITSR3 Ēś╣ņØĆ INT-ITSF1 ļ░Å ThripsITSR3)ļź╝ 10 pmolņö® ļ░śņØæņĢĪņŚÉ ļäŻĻ│Ā ņĀ£ņĪ░ĒÜīņé¼ņØś ņŗżĒŚśļ░®ļ▓ĢņŚÉ ļö░ļØ╝ SuPrimeScript RT-PCR premix (GeNetBio, Nonsan, Korea)ļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ļ░śņØæņŗ£ņ╝░ļŗż. ĻĄÉņ░©ļ░śņØæņØä ĒÖĢņØĖĒĢśĻĖ░ ņ£äĒĢ┤ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłņŚÉņä£ ņČöņČ£ĒĢ£ ĒĢĄņé░ņŚÉ ļīĆļ¦īņ┤Øņ▒äļ▓īļĀł ņ¦äļŗ©ņØä ņ£äĒĢ£ ĒöäļØ╝ņØ┤ļ©Ė(INT-ITSF1 ļ░Å ThripsITSR3)ļź╝ ļäŻņŚłĻ│Ā, ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłņŚÉņä£ ņČöņČ£ĒĢ£ ĒĢĄņé░ņŚÉļŖö ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ņ¦äļŗ©ņØä ņ£äĒĢ£ ĒöäļØ╝ņØ┤ļ©Ė(OCC-ITS2F6ņÖĆ ThripsITSR3)ļź╝ ļäŻņ¢┤ RT-PCRņØä ņłśĒ¢ēĒĢśņśĆļŗż. RT-PCR ņĪ░Ļ▒┤ņØĆ 50┬░CņŚÉņä£ 50ļČä(1ĒÜī) ļÅÖņĢł ņŚŁņĀäņé¼ ļ░śņØæņŚÉ ņØ┤ņ¢┤ 95┬░CņŚÉņä£ 2ļČä(1ĒÜī), 95┬░C 15ņ┤ł, 55┬░C 30ņ┤ł, 72┬░C, 50ņ┤ł(35ĒÜī ļ░śļ│Ą), 72┬░CņŚÉņä£ 5ļČä(1ĒÜī)ņØ┤ņŚłļŗż. ņĀäĻĖ░ņśüļÅÖņØĆ 0.5├ŚTBE ņÖäņČ®ņĢĪņÖĆ 1.0% ņĢäĻ░ĆļĪ£ņŖż ņĀżņŚÉņä£ ņŗżņŗ£ĒĢśņśĆĻ│Ā, DNA ņé¼ņØ┤ņ”ł ļ¦łņ╗żļĪ£ HiQ 100 bp plus DNA ladder (BioD, Gwangmyeong, Korea)ļź╝ ņØ┤ņÜ®ĒĢśņśĆļŗż. PCR ņ”ØĒÅŁ ņé░ļ¼╝ņØĆ 110ļ│╝ĒŖĖņŚÉņä£ 50ļČä ļÅÖņĢł ņĀäĻĖ░ņśüļÅÖ Ēøä UV transilluminatorļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ Ļ▓░Ļ│╝ļź╝ ĒÖĢņØĖĒĢśņśĆļŗż.
2019ļģä ĻĄŁĒÖö ņŻ╝ņÜö ņ×¼ļ░░ņ¦ĆņŚŁ ņżæ ĒĢśļéśņØĖ Ļ▓Įļé© ņ░ĮņøÉņŗ£ ņåīņ×¼ ņŗ£ņäżĒĢśņÜ░ņŖżņŚÉņä£ ņ×¼ļ░░ļÉśļŖö ņŖżĒāĀļŗżļō£ ĻĄŁĒÖö(ļ░▒ļ¦ł ĒÆłņóģ)ļź╝ ņĪ░ņé¼ĒĢ£ Ļ▓░Ļ│╝, 2ņøöŌĆÆ3ņøö ĻĄŁĒÖö ņĀĢņŗØ ņØ┤Ēøä 4ņøöĻ▓Į ņ┤Øņ▒äļ▓īļĀłĻ░Ć ĒĢśņÜ░ņŖż ņŗ£ņäż ļé┤ļĪ£ ņ£Āņ×ģļÉśņ¢┤ ņ┤Øņ▒äļ▓īļĀłĻ░Ć TSWV Ļ░ÉņŚ╝ ĻĄŁĒÖöņŚÉņä£ TSWVļź╝ ņ▓┤ļé┤ ļ│┤ļÅģ Ēøä ņ¦ĆņåŹņĀüņ£╝ļĪ£ TSWV Ļ░ÉņŚ╝ņØä ĒÖĢņé░ņŗ£ĒéżļŖö Ļ▓āņ£╝ļĪ£ ĒÖĢņØĖļÉśņŚłļŗż. 5ņøöļČĆĒä░ ņ£ĪņĢł ĒÖĢņØĖņØ┤ Ļ░ĆļŖźĒĢ£ TSWV ļ│æņ¦ĢņØ┤ ļéśĒāĆļéśĻĖ░ ņŗ£ņ×æĒ¢łļŗż. ļ│æņ¦Ģ ņ┤łĻĖ░ņŚÉļŖö Ļ░ÉņŚ╝ ĻĄŁĒÖö ņ×ÄņØ┤ ĒŗĆņ¢┤ņ¦ĆĻ▒░ļéś ņżäĻĖ░Ļ░Ć Ē£śņ¢┤ņ¦ĆļŖö ļ│æņ¦ĢņØ┤ ļéśĒāĆļé¼ņ£╝ļ®░, ļ│æņØ┤ ņ¦äņĀäļÉśļ®┤ņä£ ņāüņŚĮņŚÉ Ļ┤┤ņé¼ļ░śņĀÉĻ│╝ ņżäĻĖ░ Ļ┤┤ņé¼Ļ░Ć ļéśĒāĆļéś Ļ┤┤ņé¼ļÉ£ ļ░®Ē¢źņ£╝ļĪ£ ņŗØļ¼╝ņ▓┤Ļ░Ć ĻĖ░ņÜĖņ¦ĆļŖö ļ│æņ¦ĢņØ┤ Ļ┤Ćņ░░ļÉśņŚłļŗż. Ļ░ÉņŚ╝ ņØ┤Ēøä 2Ļ░£ņøö ņØ┤ļé┤ņŚÉ ņĀĢļŗ©ļČĆ ņżäĻĖ░Ļ░Ć Ļ┤┤ņé¼ļÉśļ®┤ņä£ ņāüņŚĮņØ┤ Ļ│Āņé¼ļÉśļŖö ņ┤łĻĖ░ ļ│æņ¦ĢņØ┤ ļéśĒāĆļé¼Ļ│Ā ņØ┤Ēøä ņŗØļ¼╝ņ▓┤Ļ░Ć ņĀäņ▓┤Ļ░Ć Ļ│Āņé¼Ē¢łļŗż(Fig. 1A). TSWVņŚÉ ņØśĒĢ£ Ļ░ÉņŚ╝ņŚ¼ļČĆļź╝ ĒÖĢņØĖĒĢśĻĖ░ ņ£äĒĢ┤ Ļ░ÉņŚ╝ ņ×ÄņØä ļ¦łņćäĒĢ£ ņĪ░ņ”ÖņĢĪņŚÉ Im-munoStrip (Agdia)ļź╝ ļŗ┤Ļ░Ć ĒÖĢņØĖĒĢ£ Ļ▓░Ļ│╝, TSWV ĒĢŁņ▓┤-ĻĖł ņ×ģņ×ÉĻ░Ć TSWVņÖĆ ļ░śņØæĒĢ£ ļČēņØĆ ņäĀņØ┤ 2Ļ░£Ļ░Ć ļéśĒāĆļéś TSWV Ļ░ÉņŚ╝ņØ┤ ĒÖĢņ¦äļÉśņŚłļŗż(Fig. 1B). ņŚŁĒĢÖ ņĪ░ņé¼ Ļ▓░Ļ│╝, ĻĄŁĒÖö ņŗ£ņäżĒĢśņÜ░ņŖżņØś ņĖĪņ░Į ņŻ╝ļ│ĆņŚÉ ņŗ¼ņØĆ ĻĄŁĒÖöņŚÉņä£ Ļ░Ćņן ļ©╝ņĀĆ TSWV ņ”ØņāüņØ┤ ļéśĒāĆļéśļŖö Ļ▓āņ£╝ļĪ£ ĒÖĢņØĖļÉśņŚłņ£╝ļ®░ ņ┤Øņ▒äļ▓īļĀłņØś ņ”ØĻ░ĆņÖĆ ĒĢ©Ļ╗ś ņŗ£ņäżĒĢśņÜ░ņŖż Ļ░Ćņן ņĢłņ¬ĮņØś ĻĄŁĒÖöĻ╣īņ¦Ć ĒÖĢņé░ļÉśļŖö Ļ▓āņ£╝ļĪ£ ĒÖĢņØĖļÉśņŚłļŗż(data not shown).
TSWVņØś ĒŖ╣ņØ┤ņĀüņØĖ ĒöäļØ╝ņØ┤ļ©Ėļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ Ļ│ĀņČöņŚÉņä£ ņ”ØņŗØļÉ£ TSWVļź╝ ņ¦äļŗ©ĒĢ£ Ļ▓░Ļ│╝ 777 bpĻ░Ć ĒÖĢņØĖļÉśņŚłļŗż(Supplementary Fig. 1A). ļśÉĒĢ£ ĻĄŁĒÖö ņŗ£ņäżĒĢśņÜ░ņŖż ņŻ╝ļ│ĆņŚÉņä£ ņ▒äņ¦æĒĢ£ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ļ░Å ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłļź╝ ĒśĢĒā£ņĀüņ£╝ļĪ£ ĻĄ¼ļČäĒĢ£ Ēøä Ļ░ü 10ļ¦łļ”¼ņö®ņØä ļ¬©ņĢäņä£ ĒĢĄņé░ņØä ņČöņČ£ĒĢ£ Ēøä ĒŖ╣ņØ┤ ĒöäļØ╝ņØ┤ļ©Ėļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ RT-PCRņØä ņłśĒ¢ēĒĢ£ Ļ▓░Ļ│╝, ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłļŖö 287 bp, ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłļŖö 367 bpņØś DNAĻ░Ć ĻĄÉņ░©ļ░śņØæ ņŚåņØ┤ ņ”ØĒÅŁļÉśņŚłļŗż(data not shown). ņ┤Øņ▒äļ▓īļĀł 1 ļ¦łļ”¼(0.05 mg)ņŚÉņä£ ņ┤Øņ▒äļ▓īļĀłņŚÉņä£ ņóģ ĻĄ¼ļČä ļ░Å ņ▓┤ļé┤ņŚÉ ļ│┤ļÅģĒĢśĻ│Ā ņ׳ļŖö TSWVļź╝ ļÅÖņŗ£ņ¦äļŗ©ĒĢśĻĖ░ ņ£äĒĢ┤ ĒĢĄņé░ņØä ņČöņČ£ĒĢśņśĆņ£╝ļ®░, ņĀĢļ¤ēĒĢ£ Ļ▓░Ļ│╝ 100ŌĆÆ200 ngņØś ĒĢĄņé░ņØ┤ ņČöņČ£ļÉśņŚłļŗż. 10ŌĆÆ20 ng ĒĢĄņé░ņØä ņØ┤ņÜ®ĒĢśņŚ¼ ņĢ×ņä£ ĻĖ░ņłĀĒĢ£ ņ┤Øņ▒äļ▓īļĀłņØś ņóģ ĒŖ╣ņØ┤ņĀü ĒöäļØ╝ņØ┤ļ©Ėļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ RT-PCRņØä ņłśĒ¢ēĒĢ£ Ļ▓░Ļ│╝ ņ”ØĒÅŁņé░ļ¼╝ņØ┤ ļ╣äĒŖ╣ņØ┤ņĀü ļ░śņØæ ņŚåņØ┤ ņ”ØĒÅŁļÉśņŚłļŗż(Supplementary Fig. 1B, C). ĻĄŁĒÖö ņŗ£ņäżņ×¼ļ░░ņ¦ĆņŚÉņä£ Ēżņ¦æĒĢ£ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ņä▒ņČ®ļ┐Éļ¦ī ņĢäļŗłļØ╝ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł 1ļĀ╣ ņĢĮņČ®ņŗ£ĻĖ░ņŚÉ TSWVļź╝ ĒØĪņ”Öņŗ£Ēé© Ēøä ņé¼ņ£ĪĒĢ£ 2ļĀ╣ ņĢĮņČ®Ļ│╝ ļ▓łļŹ░ĻĖ░ 1ļ¦łļ”¼ņŚÉņä£ TSWVĻ░Ć ņ¦äļŗ©ļÉśņŚłļŗż(Supplementary Fig. 2).
ņØĖņ£äņĀüņ£╝ļĪ£ TSWVļź╝ ļ│┤ļÅģņŗ£Ēé© F. occidentalis ļ░Å ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłņØś ņä▒ņČ® 1ļ¦łļ”¼ņŚÉņä£ TSWVņÖĆ ņ┤Øņ▒äļ▓īļĀł ņóģ ļÅÖņĀĢņØä ļÅÖņŗ£ ņ¦äļŗ©ĒĢ£ Ļ▓░Ļ│╝, ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ļ░Å ļīĆļ¦īņ┤Øņ▒äļ▓īļĀł ĻĄÉņ░© ļ░śņØæņØ┤ ņŚåņØ┤ TSWVņÖĆ ņ┤Øņ▒äļ▓īļĀł ņóģ ĒŖ╣ņØ┤ņĀüņØĖ ņ”ØĒÅŁ ņé░ļ¼╝ņØ┤ ĒĢ®ņä▒ņØ┤ ļÉśņ¢┤ ļÅÖņŗ£ņ¦äļŗ©ņØ┤ Ļ░ĆļŖźĒĢ£ Ļ▓āņ£╝ļĪ£ ĒīÉņĀĢļÉśņŚłļŗż(Fig. 2B, C). ĻĖ░ņĪ┤ņØś ņ┤Øņ▒äļ▓īļĀłņØś ņóģņØä ĻĄ¼ļČäĒĢśĻĖ░ ņ£äĒĢ£ ļČäņ×Éņ¦äļŗ©ņØĆ ļ»ĖĒåĀņĮśļō£ļ”¼ņĢä DNA cytochrome oxidase subunit I ļČĆļČäņØä PCRļĪ£ ņ”ØĒÅŁĒĢ£ ļŗżņØī ņĀ£ĒĢ£ĒÜ©ņåī RsaIņ£╝ļĪ£ ņĀłļŗ©ĒĢśņŚ¼ ļéśĒāĆļé£ DNA ļŗ©ĒÄĖņØś Ēī©Ēä┤ņ£╝ļĪ£ ĻĄ¼ļČäĒĢśļŖö ļ░®ļ▓ĢņØ┤ļéś(Brunner ļō▒, 2002) Ēś╣ņØĆ ribosomal ITS2 ņ£ĀņĀäņ×Éļź╝ PCRļĪ£ ņ”ØĒÅŁ Ēøä ņ┤Øņ▒äļ▓īļĀłņØś Ļ░ü ņóģļ¦łļŗż ņä£ļĪ£ ļŗżļźĖ ņĀ£ĒĢ£ĒÜ©ņåīļź╝ ņ▓śļ”¼ĒĢśņŚ¼ restriction fragment length polymorphism Ļ▓░Ļ│╝ļĪ£ ĒīÉņĀĢĒĢśļŖö ļ░®ļ▓ĢņØä ņé¼ņÜ®ĒĢśņśĆļŗż(Brunner ļō▒, 2002; TodaņÖĆ Komazaki, 2002). ņĄ£ĻĘ╝ņŚÉļŖö ļō▒ņś©ņ”ØĒÅŁļ▓Ģ(loop-mediated isothermal amplification) ļ░Å recombinase polymerase amplification ņ¦äļŗ©ļ▓ĢņØ┤ Ļ░£ļ░£ļÉśņŚłļŗż(Fekrat ļō▒, 2015; Fukuda ļō▒, 2005; Priti ļō▒, 2020; Przybylska ļō▒, 2015). ĻĘĖļ¤¼ļéś ņ▓śļ”¼ĒĢśļŖö ļŹ░ ņŗ£Ļ░äņØ┤ ļ¦ÄņØ┤ ņåīņÜöļÉśļ®░ PCR ļ░śņØæņØ┤ ļüØļé£ ĒøäņŚÉļÅä ņĀ£ĒĢ£ĒÜ©ņåīļź╝ ņČöĻ░ĆļĪ£ ņ▓śļ”¼ĒĢśĻ▒░ļéś, ņ×¼Ēśäņä▒ņØ┤ ļ¢©ņ¢┤ņ¦ĆĻ▒░ļéś, ĒöäļØ╝ņØ┤ļ©Ė ņĀ£ņ×æņŚÉ ļ│Ąņ×ĪĒĢ£ ņĀłņ░©Ļ░Ć ĒĢäņÜöĒĢśļŗżļŖö ļŗ©ņĀÉņØ┤ ņ׳ņŚłļŗż. ņØ┤ļ▓ł ņŚ░ĻĄ¼ņŚÉņä£ Ļ░£ļ░£ļÉ£ ņ┤Øņ▒äļ▓īļĀł ļ░Å TSWV ļÅÖņŗ£ ļŗżņżæ PCR ņ¦äļŗ©ļ▓ĢņØĆ ĻĄŁĒÖö ņŗ£ņäżĒĢśņÜ░ņŖżņŚÉņä£ ņÖĖļČĆ ņ£Āņ×ģ ņ┤Øņ▒äļ▓īļĀłņØś ļ░öņØ┤ļ¤¼ņŖż ļ│┤ļÅģ ņŚ¼ļČĆļź╝ ņēĮĻ│Ā ļ╣Āļź┤Ļ▓ī ņ¦äļŗ©ĒĢĀ ņłś ņ׳ĻĖ░ ļĢīļ¼ĖņŚÉ ļ░öņØ┤ļ¤¼ņŖżļ│æ ņ£Āņ×ģ ļ░Å ĒÖĢņé░ņØä ņśłņĖĪĒĢśļŖö ļŹ░ ĒÖ£ņÜ®ĒĢĀ ņłś ņ׳ņØä Ļ▓āņ£╝ļĪ£ ĒīÉļŗ©ļÉ£ļŗż(Jung ļō▒ 2019; Kang ļō▒, 2012).
ņ┤Øņ▒äļ▓īļĀł 1ļ¦łļ”¼ņŚÉņä£ TSWV ļ│┤ļÅģ ņŚ¼ļČĆ ļ░Å ņóģ ļÅÖņĀĢņØ┤ Ļ░ĆļŖźĒĢ£ Ļ▓░Ļ│╝ņØś ĒÜ©ņÜ®ņä▒ņØä ņĪ░ņé¼ĒĢśĻĖ░ ņ£äĒĢśņŚ¼, ĻĄŁĒÖö ņ×¼ļ░░ ĒĢśņÜ░ņŖżņŚÉņä£ ņ┤Øņ▒äļ▓īļĀłņÖĆ TSWVļź╝ ļÅÖņŗ£ ņ¦äļŗ©ņØä ņŗżņŗ£ĒĢśņśĆļŗż. ņØ┤ļ¤░ ļ¬®ņĀüņ£╝ļĪ£ ņČ®ļé© Ēā£ņĢłĻĄ░ ņåīņ×¼ ņŖżĒöäļĀłņØ┤ ĻĄŁĒÖö(ņåīĻĄŁ) ņ×¼ļ░░ ĒĢśņÜ░ņŖż ļ░Å Ļ▓Įļé© ņ░ĮņøÉņŗ£ ņåīņ×¼ ņŖżĒāĀļŗżļō£ ĻĄŁĒÖö(ļīĆĻĄŁ) ņ×¼ļ░░ ĒĢśņÜ░ņŖżņŚÉņä£ ĒāĆļØĮļ▓ĢņØä ņØ┤ņÜ®ĒĢśņŚ¼ Ļ░üĻ░ü 43, 102ļ¦łļ”¼ļź╝ ļ¼┤ņ×æņ£äļĪ£ ņ▒äņ¦æĒĢ£ ļŗżņØī ļÅÖņŗ£ņ¦äļŗ©ļ▓Ģņ£╝ļĪ£ ņóģ ļÅÖņĀĢ ļ░Å TSWV ļ│┤ļÅģ ņŚ¼ļČĆļź╝ ņĪ░ņé¼ĒĢśņśĆļŗż.
Ēā£ņĢłĻĄ░ņŚÉņä£ ņ▒äņ¦æĒĢ£ ņ┤Øņ▒äļ▓īļĀłņŚÉņä£ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłļŖö 36ļ¦łļ”¼ļĪ£ 83.7%ļź╝ ņ░©ņ¦ĆĒ¢łņ£╝ļ®░, ļÅÖņĀĢļÉ£ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłņŚÉņä£ 36ļ¦łļ”¼ ņżæ 27ļ¦łļ”¼ņŚÉņä£ TSWVĻ░Ć ņ¦äļŗ©ļÉśņ¢┤, ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłņØś TSWV ļ│┤ļÅģļźĀņØĆ 72.9%ļĪ£ ņĪ░ņé¼ļÉśņŚłļŗż(Table 2). ņóģ ļÅÖņĀĢ Ļ▓░Ļ│╝ ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłļŖö 1ļ¦łļ”¼ļĪ£ ĒīÉņĀĢļÉśņŚłņ£╝ļ®░, TSWVļŖö ļ│┤ļÅģĒĢśĻ│Ā ņ׳ņ¦Ć ņĢŖņĢśņ£╝ļ®░, ļ»ĖļÅÖņĀĢ ņ┤Øņ▒äļ▓īļĀłļŖö 43ļ¦łļ”¼ ņżæ 6ļ¦łļ”¼ņØ┤ņŚłņ£╝ļ®░, TSWV ļ│┤ļÅģļźĀņØĆ 100% (6ļ¦łļ”¼ ņżæ 6ļ¦łļ”¼)ļĪ£ ņĪ░ņé¼ļÉśņŚłļŗż(Table 2).
Ļ▓Įļé© ņ░ĮņøÉņŗ£ņŚÉņä£ ņ▒äņ¦æĒĢ£ ņ┤Øņ▒äļ▓īļĀł ņóģ ļÅÖņĀĢ ļ░Å TSWV ļ│┤ļÅģļźĀņØä ņĪ░ņé¼ĒĢ£ Ļ▓░Ļ│╝, 102 ļ¦łļ”¼ ņżæ 94ļ¦łļ”¼(92.2%)Ļ░Ć ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłļĪ£ ĒÖĢņØĖļÉśņŚłņ£╝ļ®░, 94ļ¦łļ”¼ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ņżæ 79 ļ¦łļ”¼ņŚÉņä£ TSWVĻ░Ć Ļ▓ĆņČ£ļÉśņ¢┤ TSWV ļ│┤ļÅģļźĀņØĆ 84.0%ļĪ£ ņĪ░ņé¼ļÉśņŚłļŗż(Table 2). ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłļź╝ ļÅÖņĀĢĒĢ£ Ļ▓░Ļ│╝ 102ļ¦łļ”¼ ņżæ 4ļ¦łļ”¼(3.9%)ņØ┤ņŚłņ£╝ļ®░ TSWV ļ│┤ļÅģļźĀņØĆ 50%ļĪ£ ņĪ░ņé¼ļÉśņŚłļŗż(Table 2). ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ļ░Å ļīĆļ¦īņ┤Øņ▒äļ▓īļĀł ņØ┤ņÖĖņŚÉ ļŗżļźĖ ņóģņØś ņ┤Øņ▒äļ▓īļĀłļŖö 102ļ¦łļ”¼ ņżæ 4ļ¦łļ”¼(3.0%) ņØ┤ņŚłņ£╝ļ®░, TSWV ļ│┤ļÅģļźĀņØĆ 75%ļĪ£ ņĪ░ņé¼ļÉśņŚłļŗż. ņĪ░ņé¼ļÉ£ ņé¼ņŗżņØä ņóģĒĢ®ĒĢ┤ļ│╝ ļĢī, ĻĄŁĒÖö ņŗ£ņäż ĒĢśņÜ░ņŖżņŚÉņä£ ņÜ░ņĀÉņóģņØĆ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłņØ┤ļ®░ 75.0ŌĆÆ84.0% TSWV ļ│┤ļÅģļźĀņØä Ļ░Ćņ¦ĆĻ│Ā ņ׳ņ¢┤ TSWV Ļ░ÉņŚ╝ ļ░Å ņĀäĒīīņŚÉ Ļ░Ćņן Ēü░ ņÜöņØĖņ×äņØä ĒīÉļŗ©ļÉ£ļŗż. ĒØźļ»ĖļĪŁĻ▓īļÅä ļ╣äļĪØ ņ┤Øņ▒äļ▓īļĀł ņóģ ļÅÖņĀĢņØĆ ĒÖĢņØĖĒĢĀ ņłś ņŚåņŚłņ£╝ļéś ļ»ĖļÅÖņĀĢ ņ┤Øņ▒äļ▓īļĀłņŚÉņä£ TSWV ļ│┤ļÅģļźĀņØĆ ņāüļŗ╣Ē׳ ļåÆņØĆ ņłśņżĆņ£╝ļĪ£ ņĪ░ņé¼ļÉśņŚłļŗż. TSWVļź╝ ļ¦żĻ░£ĒĢĀ ņłś ņ׳ļŗżĻ│Ā ņĢīļĀżņ¦ä ņśżņØ┤ņ┤Øņ▒äļ▓īļĀł ņŚ¼ļČĆļź╝ ļ»ĖļÅÖņĀĢļÉ£ ņ┤Øņ▒äļ▓īļĀłļź╝ Ēśäļ»ĖĻ▓ĮņŚÉņä£ ĒśĢĒā£ĒĢÖņĀü ļČäļźśļź╝ ĒĢśņśĆņ£╝ļéś ņśżņØ┤ņ┤Øņ▒ä ļ▓īļĀłļŖö ĒÖĢņØĖļÉśņ¦Ć ņĢŖņĢśļŗż(data not shown). ĻĘĖ ņÖĖ ĻĄŁĒÖöņŚÉņä£ ņä£ņŗØĒĢśļŖö ņ┤Øņ▒äļ▓īļĀłļŖö Ēīīņ┤Øņ▒äļ▓īļĀł, ĒĢśņÖĆņØ┤ņ┤Øņ▒äļ▓īļĀł, ļ»Ėļéśļ”¼ņ┤Øņ▒äļ▓īļĀł ļō▒ 10ņŚ¼ņóģņØ┤ ĒÖĢņØĖļÉśĻ│Ā ņ׳ņ£╝ļéś TSWV ļ│┤ļÅģ ņŚ¼ļČĆ ļō▒ņŚÉ ļīĆĒĢśņŚ¼ ļ®┤ļ░ĆĒĢ£ ņĪ░ņé¼Ļ░Ć ĒĢäņÜöĒĢśļŗżĻ│Ā ĒīÉļŗ©ļÉ£ļŗż(van de Wetering, 1999).
ņØ┤ļ▓ł ņŚ░ĻĄ¼ļŖö ĻĄŁĒÖöņŚÉņä£ ļ¼ĖņĀ£ļÉśļŖö ņ┤Øņ▒äļ▓īļĀłņØś ņóģ ļÅÖņĀĢ ļ░Å ļ│┤ļÅģ ļ░öņØ┤ļ¤¼ņŖżņØĖ ĒåĀļ¦łĒåĀļ░śņĀÉņ£äņĪ░ļ░öņØ┤ļ¤¼ņŖż(Tomato spotted wilt virus, TSWV)ļź╝ ļÅÖņŗ£ņŚÉ ĒÖĢņØĖĒĢĀ ņłś ņ׳ļŖö ņ¦äļŗ©ļ░®ļ▓ĢņØä Ļ░£ļ░£ĒĢśņśĆļŗż. ņØ┤ļŖö ņ┤Øņ▒äļ▓īļĀł 1ļ¦łļ”¼ņŚÉņä£ ņČöņČ£ĒĢ£ ĒĢĄņé░ņŚÉ ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀł ļ░Å ļīĆļ¦īņ┤Øņ▒äļ▓īļĀłņØś ITS2 ļČĆļČäņŚÉ ĒŖ╣ņØ┤ņĀüņØĖ ĒöäļØ╝ņØ┤ļ©ĖņÖĆ TSWV ņÖĖĒö╝ļŗ©ļ░▒ņ¦ł(N) ņ£ĀņĀäņ×É ĒŖ╣ņØ┤ņĀüņØĖ ĒöäļØ╝ņØ┤ļ©Ėļź╝ ļÅÖņŗ£ņŚÉ ļäŻņ¢┤ reverse tran scriptionŌĆÆpolymerase chain reactionņØä ņłśĒ¢ēĒĢśņŚ¼ DNAļź╝ ņ”ØĒÅŁņŗ£ĒéżļŖö ļ░®ļ▓Ģņ£╝ļĪ£ ņĀäĻĖ░ņśüļÅÖĒĢśņŚ¼ Ļ░üĻ░ü 287, 367, 777 bpņØś DNA ļŗ©ĒÄĖņØś Ēü¼ĻĖ░ļź╝ ļ╣äĻĄÉĒĢ©ņ£╝ļĪ£ņŹ© ņ┤Øņ▒äļ▓īļĀłņØś ņóģ ļÅÖņĀĢ ļ░Å ņ┤Øņ▒äļ▓īļĀłņØś TSWV ļ│┤ļÅģ ņŚ¼ļČĆļź╝ ļÅÖņŗ£ņŚÉ ĒÖĢņØĖĒĢĀ ņłś ņ׳ļŗż. ņČ®ņ▓Łļé©ļÅä Ēā£ņĢł ļ░Å Ļ▓Įņāüļé©ļÅä ņ░ĮņøÉņØś ĻĄŁĒÖö ņŗ£ņäżĒĢśņÜ░ņŖżņŚÉņä£ ņ┤Øņ▒äļ▓īļĀłļź╝ Ēżņ¦æĒĢśņŚ¼ ņ┤Øņ▒äļ▓īļĀł ņÜ░ņĀÉņóģĻ│╝ ņ┤Øņ▒äļ▓īļĀłņØś TSWV ļ│┤ļÅģņ£©ņØä ņĪ░ņé¼ĒĢ£ Ļ▓░Ļ│╝, Ēā£ņĢłņØś ĻĄŁĒÖö ņŗ£ņäżĒĢśņÜ░ņŖżņŚÉņä£ļŖö ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłĻ░Ć 83.7%ļĪ£ ņÜ░ņĀÉĒĢśĻ│Ā ņ׳ņ£╝ļ®░ ņ▒äņ¦æļÉ£ ņ┤Øņ▒äļ▓īļĀł ņżæ 72.9%Ļ░Ć TSWVļź╝ ļ│┤ļÅģĒĢśĻ│Ā ņ׳ņŚłņ£╝ļ®░, ņ░ĮņøÉņŚÉņä£ļŖö ĻĮāļģĖļ×æņ┤Øņ▒äļ▓īļĀłĻ░Ć 92.2%ļź╝ ņ░©ņ¦ĆĒĢśĻ│Ā ņ׳ņ£╝ļ®░ 84.0%ņØś ņ┤Øņ▒äļ▓īļĀłņŚÉņä£ TSWVĻ░Ć ņ¦äļŗ©ļÉśņŚłļŗż. ņØ┤ļ¤¼ĒĢ£ Ļ▓░Ļ│╝ļŖö Frankliniella occidentalisĻ░Ć ņÜ░ņĀÉņóģņØ┤ļ®░ ņś©ņŗżņØś ĻĄŁĒÖö ņŗØļ¼╝ņŚÉņä£ TSWVņØś ņĀäļ░śņŚÉ ņżæņÜöĒĢ£ ņŚŁĒĢĀņØä ĒĢ£ļŗżļŖö Ļ▓āņØä ĒÖĢņØĖĒĢ┤ņżĆļŗż. ņØ┤ļ▓ł ņŚ░ĻĄ¼ļŖö ĻĄŁĒÖö ņŗ£ņäżĒĢśņÜ░ņŖżņŚÉņä£ ņ┤Øņ▒äļ▓īļĀłļź╝ ĒåĄĒĢ£ TSWVņØś ņŗ£ņäżĒĢśņÜ░ņŖżļé┤ ņ£Āņ×ģņŗ£ĻĖ░ ļ░Å ĒÖĢņé░ Ļ▓ĮļĪ£ ļō▒ ļ░öņØ┤ļ¤¼ņŖżņØś ņŚŁĒĢÖņŚ░ĻĄ¼ļź╝ ņ£äĒĢ£ Ļ░äĒÄĖņ¦äļŗ©ļ▓Ģņ£╝ļĪ£ ĒÖ£ņÜ® Ļ░ĆļŖźĒĢ©ņØä ņśłņŗ£ĒĢ┤ņżĆļŗż.
Supplementary materials are available at Research in Plant Disease website (http://www.online-rpd.org/).
Acknowledgments
This work was supported by a grant from the Basic Research Program (Project no. PJ01359101) of National Institute of Horticultural and Herbal Science, RDA, South Korea.
Fig.┬Ā1
Symptoms of chrysanthemum plants infected with TSWV and diagnostic results of TSWV in infected chrysanthemum plants. Typical symptoms of chrysanthemum plants infected with TSWV were presented as arrows at different developmental stages. (A) At the early infection stage, most of the TSWV-infected chrysanthemum plants exhibited chlorosis or chlorosis and one-sided necrosis on leaves. At the mid-term infection stage, most of the infected chrysanthemum plants developed necrotic ringspots and necrosis on leaves, severe necrosis on stems, stunting, and terminal buds with signs of necrosis. At the late infection stage, most of the infected chrysanthemum plants exhibited systemically severe necrosis, stunting and drooping leaves. Some plant death was commonly observed. (B) Positive reaction to TSWV in the TSWV-ImmunoStrip (Agdia), suggesting that the chrysanthemum plants authentically were infected with TSWV.

Fig.┬Ā2
Schematic represent of simultaneous diagnostic method for identification of thrips species and TSWV in an individual thrips. (A) Total nucleic acid was extracted from one thrips using DNA/RNA extraction kit. Then, reverse transcription-polymerase chain reaction (RT-PCR) analysis was used for identification of thrips species and determination of thrips carrying TSWV, based on different sizes of amplified RT-PCR products in agarose gel. (B) Results of simultaneous identification of F. occidentalis and the acquired TSWV with primers specific to TSWV and F. occidentalis (left) and with primers specific to TSWV and F. intonsa (right). Lane M, HiQ 100 bp plus DNA ladder; lane 1, TSWV-infected pepper leaf; lane 2, F. occidentalis maintained in the lab; lanes 3-5, F. occidentalis that acquire TSWV. (C) Results of simultaneous identification of F. intonsa and the acquired TSWV with primers specific to TSWV and F. intonsa (left) and with primers specific to TSWV and F. occidentalis (right). Lane M, HiQ 100 bp plus DNA ladder; lane 1, TSWV-infected pepper leaf; lane 2, F. intonsa maintained in the lab; lanes 3-5, F. intonsa that acquire TSWV. Molecular mass of HiQ 100 bp plus DNA ladder was indicated in the left and the amplified RT-PCR products were indicated as narrows.
Table┬Ā1
Primer sequences used in this study
| Target species | Primer sequence | Size (bp) | Reference |
|---|---|---|---|
| TSWV | TSWV-NCP-For: 5ŌĆÖ-ATGTCTAAGGTTAAGCTCACTAAGGAA-3ŌĆÖ | 777 | Yoon et al. (2014) |
| TSWV-NCP-Rev: 5ŌĆÖ-TTAAGCAAGTTCTGCAAGTATTGCCTG-3ŌĆÖ | |||
| F. occidentalis | OCC-ITS2F6: 5ŌĆÖ-GGTCGCTTCACCGCTTCCCG-3ŌĆÖ | 287 | Nakahara and Minoura (2015) |
| ThripsITS2R3: 5ŌĆÖ-CTCTCCTGAACWGAGGTCG-3ŌĆÖ | |||
| F. intonsa | INT-ITS2F1: 5ŌĆÖ-GACCAGACTGTTCCGAGA-3ŌĆÖ | 367 | |
| ThripsITS2R3: 5ŌĆÖ-CTCTCCTGAACWGAGGTCG-3ŌĆÖ |
Table┬Ā2
Greenhouse survey for thrips collected from greenhouses in Taean and Changwon, South Korea
References
Bandla, M. D., Westcot, D. M., Chenault, K. D., Ullman, D. E., German, T. L. and Sherwood, J. L. 1994. Use of monoclonal antibody to the nonstructural protein encoded by the small RNA of Tomato spotted wilt tospovirus to identify viruliferous thrips. Phytopathology 84: 1427-1431.

Boonham, N., Smith, P., Walsh, K., Tame, J., Morris, J., Spence, N. et al. 2002. The detection of Tomato spotted wilt virus (TSWV) in individual thrips using real time fluorescent RT-PCR (TaqMan). J. Virol. Methods 101: 37-48.


Brunner, P. C., Fleming, C. and Frey, J. E. 2002. A molecular identification key for economically important thrips species (Thysanoptera: Thripidae) using direct sequencing and a PCR-RFLP-based approach. Agric. For. Entomol 4: 127-136.

Cho, J. J., Mau, R. F. L., German, T. L., Hartmann, R. W., Yudin, L. S., Gonsalves, D. et al. 1989. A multidisciplinary approach to management of Tomato spotted wilt virus in Hawaii. Plant Dis 73: 375-383.

Chung, B. N., Pak, H. S., Jung, J. A. and Kim, J. S. 2006. Occurrence of Tomato spotted wilt virus in Chrysanthemum (Dendranthema grandiflorum) in Korea. Plant Pahtol. J 22: 230-234.

Demirozer, O., Tyler-Julian, K., Funderburk, J., Leppla, N. and Reitz, S. 2012. Frankliniella occidentalis (Pergande) integrated pest management programs for fruiting vegetables in Florida. Pest Manag. Sci 68: 1537-1545.


Fekrat, L., Aghl, M. and Tahan, V. 2015. Application of the LAMP assay as a diagnostic technique for rapid identification of Thrips tabaci (Thysanoptera: Thripidae). J. Econ. Entomol 108: 1337-1343.



Fukuda, S., Ichikawa, K. and Ohya, T. 2005. Identification of the thrips by the LAMP method. Kanto Tokai Hokuriku Agric. Res. Inf 2: 192-193. (In Japanese)
Han, M. J., Kim, I. S., Ahn, S. B., Lee, M. L., Hong, K. J., Lee, G. H. et al. 1998. Distribution and host plants of recently introduced western flower thrips, Frankliniella occidentalis (Pergande) (Thysanoptera: Thripidae) in Korea. RDA J. Crop Prot 40: 83-88. (In Korean)
Hyun, J.-W., Hwang, R.-Y., Lee, K.-S., Song, J.-H., Yi, P.-H., Kwon, H.-M. et al. 2012. Seasonal occurrence of yellow tea thrips, Scirtothrips dorsalis Hood (Thysanoptera: Thripidae) in citrus orchards and its damage symptoms on citrus fruits. Korean J. Appl. Entomol 51: 1-7. (In Korean)

Inoue, T., Sakurai, T., Murai, T. and Maeda, T. 2004. Specificity of accumulation and transmission of Tomato spotted wilt virus (TSWV) in two genera, Frankliniella and Thrips (Thysanoptera: Thripidae). Bull. Entomol. Res 94: 501-507.


Jung, C. Y., Jeong, D. H., Park, H. W., Kim, H. J., Jeon, K. S. and Yoon, J. B. 2019. Molecular identification of thrips in two medicinal crops, Cnidium officinale Makino and Ligusticum chuanxiong Hort. Korean J. Med. Crop Sci 27: 17-23. (In Korean)

Kang, T. J., Ahn, S. J., An, T. J., Cho, M. R., Jeon, H. Y. and Jung, J. A. 2012. Thrips in medicinal crops in Korea: identification and their damages. Korean J. Med. Crop Sci 20: 487-492. (In Korean)

Kim, J.-H., Choi, G.-S., Kim, J.-S. and Choi, J.-K. 2004. Characterization of Tomato spotted wilt virus from Paprika in Korea. Plant Pathol. J 20: 297-301.

Lee, G. S. 1996. Seasonal occurrence of Thrips palmi Karny (Thysanoptera: Thripidae) in Cheju Island. M.S. thesis. Seoul National University; Seoul, Korea: 76.(In Korean)
Lewis, T. ed. by T. Lewis, 1997. Pest thrips in perspective. In: Thrips as Crop Pests, CAB International, Wallingford, UK. pp. 1-13.
2020. Production of Flower Crops in 2019 in South Korea. Ministry of Agriculture, Food and Rural Affairs, Sejong, Korea. pp. 422 pp.
Maris, P. 2004. Evaluation of thrips resistance in pepper to control Tomato spotted wilt virus infection. Ph.D. thesis. Wageningen University; Wageningen, The Netherlands: 120.
Nakahara, S. and Minoura, K. 2015. Identification of four thrips species (Thysanoptera: Thripidae) by multiplex polymerase chain reaction. Res. Bull. Plant Prot. Jpn 51: 37-42.
Okazaki, S. and Sakurai, T. 2005. Efficiencies of transmission of Tomato spotted wilt virus by Frankliniella occidentalis and Frankliniella intonsa collected in green bell pepper fields in Oita Prefecture. Kyushu Plant Prot. Res 51: 60-63. (In Japanese)

Okuda, S., Okuda, M., Matsuura, S., Okazaki, S. and Iwai, H. 2013. Competence of Frankliniella occidentalis and Frankliniella intonsa strains as vectors for Chrysanthemum stem necrosis virus. Eur. J. Plant Pathol 136: 355-362.

Park, J.-D., Kim, D.-I. and Kim, S.-G. 2002. Seasonal occurrence and damaged aspects of Frankliniella occidentalis Pergande (Thysanoptera: Thripidae) by cultural environments and varieties of chrysanthemum. Korean J. Appl. Entomol 41: 177-181. (In Korean)
Parrella, G., Gognalons, P., Gebre-Selassi├©, K., Vovlas, C. and Marchoux, G. 2003. An update of the host range of Tomato spotted wilt virus. J. Plant Pathol 85: 227-264.
Priti, J, angra, S., Baranwal, V. K., Dietzgen, R. G. and Ghosh, A. 2020. A rapid field-based assay using recombinase polymerase amplification for identification of Thrips palmi, a vector of tospoviruses. J. Pest Sci Online publication. https://doi.org/10.1007/s10340-020-01284-w

Przybylska, A., Fiedler, ┼╗., Kucharczyk, H. and St─Öplowska, A. 2015. Detection of the quarantine species Thrips palmi by loop-mediated isothermal amplification. PLoS ONE 10: e0122033.



Schmidt, M. E. and Frey, J. E. 1995. Monitoring of the western flower thrips Frankliniella occidentalis in greenhouses. Meded. Rijksuniv. Gent. Fak. Landbouwkd. Toegep. Biol. Wet 60: 847-850.
Toda, S. and Komazaki, S. 2002. Identification of thrips species (Thysanoptera: Thripidae) on Japanese fruit trees by polymerase chain reaction and restriction fragment length polymorphism of the ribosomal ITS2 region. Bull. Entomol. Res 92: 359-363.


Tsuda, S., Fujisawa, I., Hanada, K., Hidaka, S., Higo, K.-I, Kameya-Iwaki, M. et al. 1994. Detection of Tomato spotted wilt virus S RNA in individual thrips by reverse transcription and polymerase chain reaction. Ann. Phytopathol. Soc. Jpn 60: 99-103.

van de Wetering, F. 1999. Effects of thrips feeding on tospovirus transmission in chrysanthemum. Ph.D. thesis. Wageningen University; Wageningen, The Netherlands: 120.
Wijkamp, I., Van De Wetering, F., Goldbach, R. and Peters, D. 1996. Transmission of tomato spotted wilt virus by Frankliniella occidentalism median acquisition and inoculation access period. . Ann. Appl. Biol 129: 303-313.

Yeh, W. B., Tseng, M. J., Chang, N. T., Wu, S. Y. and Tsai, Y. S. 2015. Agronomically important thrips: development of species-specific primers in multiplex PCR and microarray assay using internal transcribed spacer 1 (ITS1) sequences for identification. Bull. Entomol. Res 105: 52-59.


Yeh, W.-B., Wu, C. S.-Y., Tseng, M.-J., Chang, N.-T. and Chen, W.-P. 2014. Multiplex PCR for the molecular identification of economically important thrips. Formosan Entomol 34: 211-225. (In Chinese)
Yoon, J.-B., Choi, S.-K., Cho, I.-S., Kwon, T.-R., Yang, C.-Y., Seo, M.-H. et al. 2020. Epidemiology of tomato spotted wilt virus in Chrysanthemum morifolium in South Korea and its management using a soil-dwelling predatory mite (Stratiolaelaps scimitus) and es-sential oils. Virus Res 289: 198128.






 PDF Links
PDF Links PubReader
PubReader ePub Link
ePub Link Full text via DOI
Full text via DOI Download Citation
Download Citation Supplement
Supplement Print
Print